可视化 DESeq2 中的数值属性
通过提供通常用于转录组分析的 DESeq2 软件包的结果,可以在图形的节点中反映数值结果。该函数可用于此目的。通过将要在图形中反映的数值(例如,)指定为参数,可以将该值分配给节点。如果命中多个基因,则参数指定如何组合多个值(默认值为 )。assign_deseq2log2FoldChangecolumnnumeric_combinemean
在这里,我们使用RNA-Seq数据集,该数据集分析了感染BK多瘤病毒的人尿路上皮细胞的转录组变化(Baker等人,2022)。从 Sequence Read Archive 获得的原始序列由nf-core处理,随后使用 和 进行分析。tximportsalmonDESeq2
library(ggkegg)
library(DESeq2)
library(org.Hs.eg.db)
library(dplyr)## The file stores DESeq() result on transcriptomic dataset deposited by Baker et al. 2022.
load("uro.deseq.res.rda")
res
#> class: DESeqDataSet
#> dim: 29744 26
#> metadata(1): version
#> assays(8): counts avgTxLength ... replaceCounts
#> replaceCooks
#> rownames(29744): A1BG A1BG-AS1 ... ZZEF1 ZZZ3
#> rowData names(27): baseMean baseVar ... maxCooks
#> replace
#> colnames(26): SRR14509882 SRR14509883 ... SRR14509906
#> SRR14509907
#>服务器托管网; colData names(27): Assay.Type AvgSpotLen ...
#> viral_infection replaceable
vinf mutate(deseq2=assign_deseq2(vinf),
padj=assign_deseq2(vinf, column="padj"),
converted_name=convert_id("hsa"))
ggraph(g, layout="manual", x=x, y=y) +
geom_edge_parallel(width=0.5, arrow = arrow(length = unit(1, 'mm')),
start_cap = square(1, 'cm'),
end_cap = square(1.5, 'cm'), aes(color=subtype_name))+
geom_node_rect(aes(fill=deseq2, filter=type=="gene"), color="black")+
ggfx::with_outer_glow(geom_node_text(aes(label=converted_name, filter=type!="group"), size=2.5), colour="white", expand=1)+
scale_fill_gradient(low="blue",high="red", name="LFC")+
theme_void()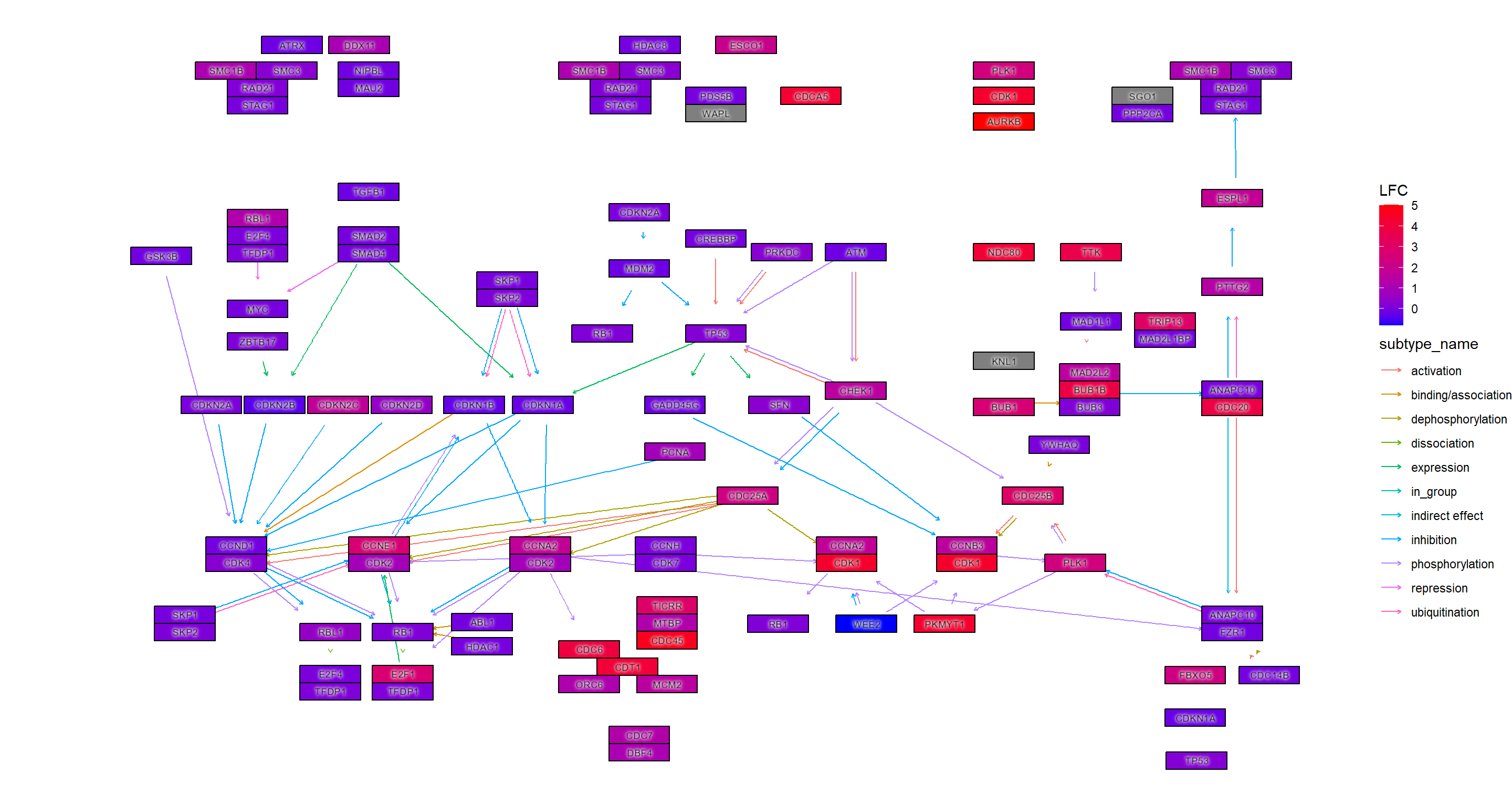
## Adjusted p-values
ggraph(g, layout="manual", x=x, y=y) +
geom_edge_parallel(width=0.5, arrow = arrow(length = unit(1, 'mm')),
start_cap = square(1, 'cm'),
end_cap = square(1.5, 'cm'), aes(color=subtype_name))+
geom_node_rect(aes(fill=padj, filter=type=="gene"), color="black")+
ggfx::with_outer_glow(geom_node_text(aes(label=converted_name, filter=type!="group"), size=2.5), colour="white", expand=1)+
scale_fill_gradient(name="padj")+
theme_void()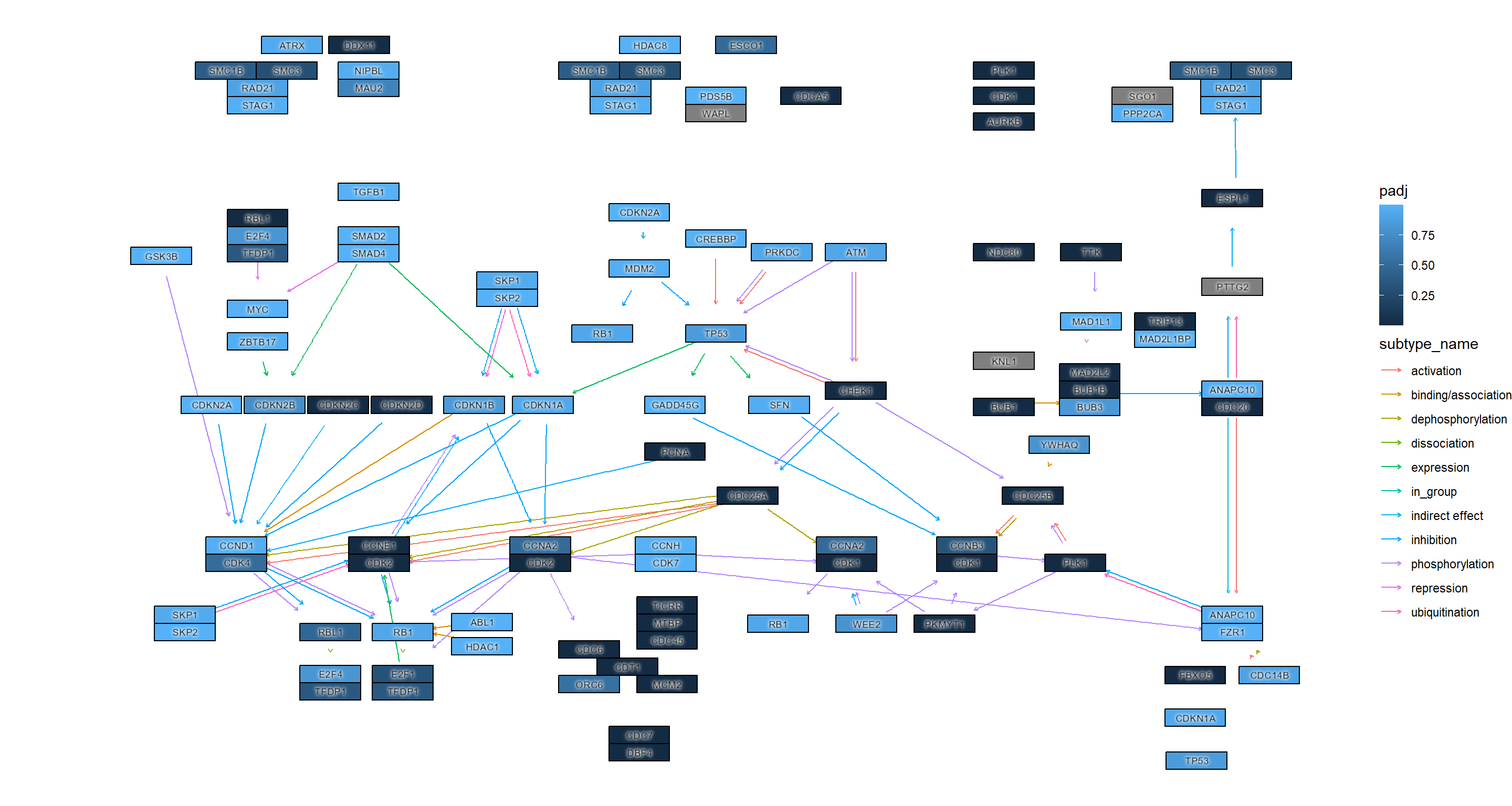
用于进一步自定义可视化ggfx
## Highlighting differentially expressed genes at adjusted p-values 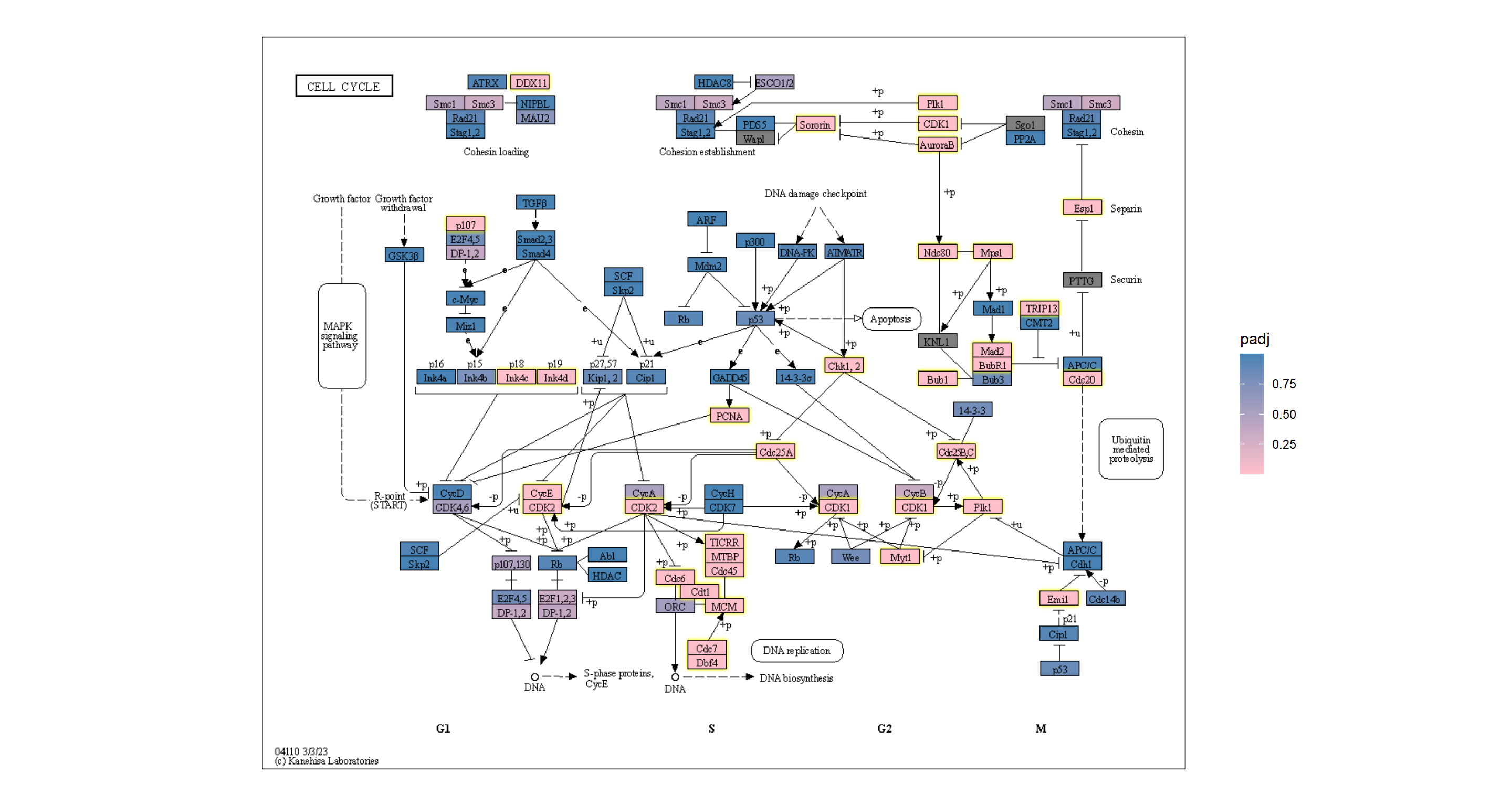
使用多个几何添加信息
您可以使用自己喜欢的几何图形及其扩展来添加信息。在此示例中,我们使用geomtextpath将 log2 折叠更改添加为轮廓,并使用Monocraft自定义字体。ggplot2
g mutate(lfc=assign_deseq2(vinf, column="log2FoldChange"))
## Make contour data
df data.frame()
df
data.frame() |> `colnames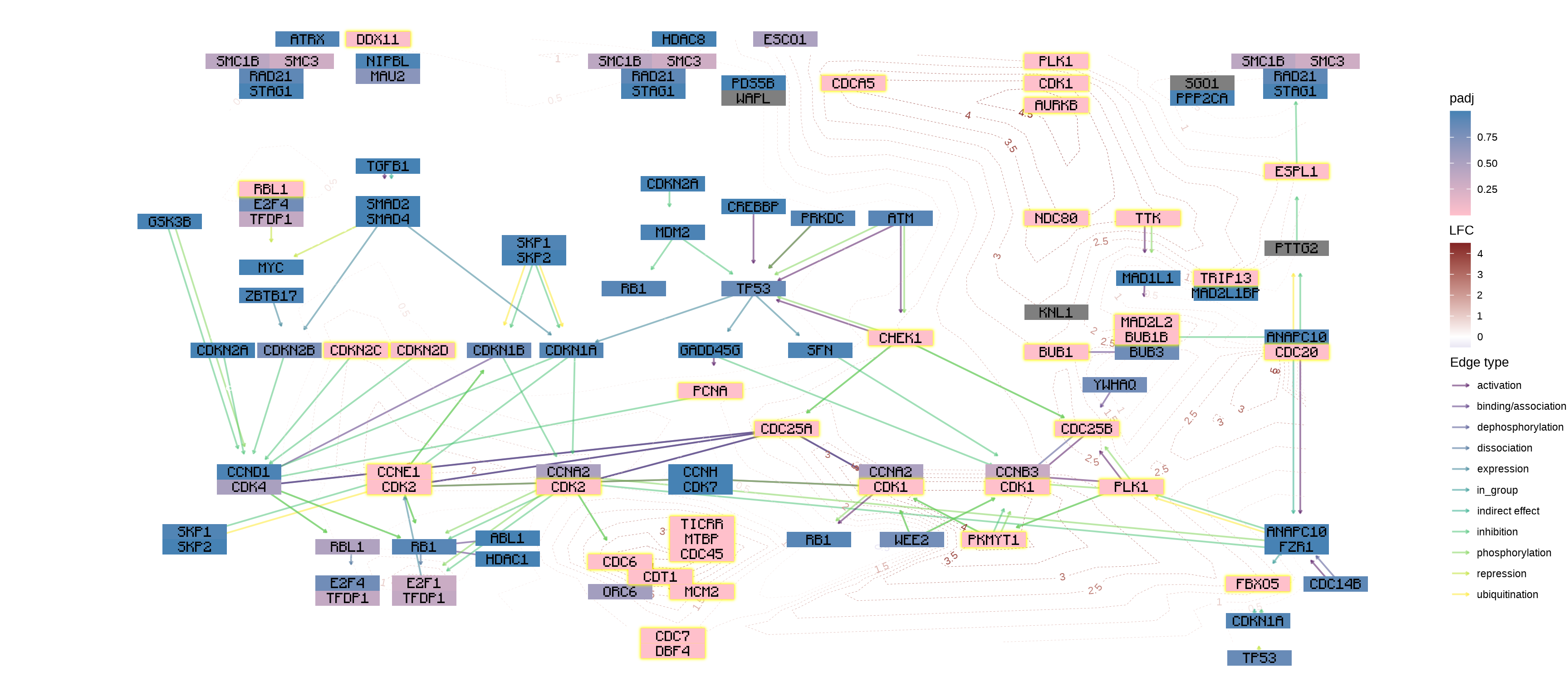
将数值积分到tbl_graph
将数值向量积分到tbl_graph
数值可以反映在节点或边表中,利用 或 函数。输入可以是命名向量,也可以是包含 id 和 value 列的 tibble。node_numericedge_numeric
vec mutate(num=node_numeric(vec))
new_g
#> # A tbl_graph: 134 nodes and 157 edges
#> #
#> # A directed acyclic multigraph with 40 components
#> #
#> # A tibble: 134 23
#> name type reaction graphics_name x y width
#>
#> 1 hsa:1029 gene CDKN2A, ARF,… 532 -218 46
#> 2 hsa:51343 gene FZR1, CDC20C… 981 -630 46
#> 3 hsa:4171 h… gene MCM2, BM28, … 553 -681 46
#> 4 hsa:23594 … gene ORC6, ORC6L.… 494 -681 46
#> 5 hsa:10393 … gene ANAPC10, APC… 981 -392 46
#> 6 hsa:10393 … gene ANAPC10, APC… 981 -613 46
#> # ℹ 128 more rows
#> # ℹ 16 more variables: height , fgcolor ,
#> # bgcolor , graphics_type , coords ,
#> # xmin , xmax , ymin , ymax ,
#> # orig.id , pathway_id , deseq2 ,
#> # padj , converted_name , lfc , num
#> #
#> # A tibble: 157 6
#> from to type subtype_name subtype_value pathway_id
#>
#> 1 118 39 GErel expression --> hsa04110
#> 2 50 61 PPrel inhibition --| hsa04110
#> 3 50 61 PPrel phosphorylation +p hsa04110
#> # ℹ 154 more rows将矩阵积分到tbl_graph
如果要在图形中反映表达式矩阵,则 和 函数可能很有用。通过指定基质和基因 ID,您可以将每个样品的数值分配给 . 分配由边连接的两个节点的总和,忽略组节点(Adnan 等人,2020年)。edge_matrixnode_matrixtbl_graphedge_matrix
mat edge_matrix(mat) |> node_matrix(mat)
new_g
#> # A tbl_graph: 134 nodes and 157 edges
#> #
#> # A directed acyclic multigraph with 40 components
#> #
#> # A tibble: 134 48
#> name type reaction graphics_name x y width
#>
#> 1 hsa:1029 gene CDKN2A, ARF,… 532 -218 46
#> 2 hsa:51343 gene FZR1, CDC20C… 981 -630 46
#> 3 hsa:4171 h… gene MCM2, BM28, … 553 -681 46
#> 4 hsa:23594 … gene ORC6, ORC6L.… 494 -681 46
#> 5 hsa:10393 … gene ANAPC10, APC… 981 -392 46
#> 6 hsa:10393 … gene ANAPC10, APC… 981 -613 46
#> # ℹ 128 more rows
#> # ℹ 41 more variables: height , fgcolor ,
#> # bgcolor , graphics_type , coords ,
#> # xmin , xmax , ymin , ymax ,
#> # orig.id , pathway_id , deseq2 ,
#> # padj , converted_name , lfc ,
#> # SRR14509882 , SRR14509883 , …
#> #
#> # A tibble: 157 34
#> from to type subtype_name subtype_value pathway_id
#>
#> 1 118 39 GErel expression --> hsa04110
#> 2 50 61 PPrel inhibition --| hsa04110
#> 3 50 61 PPrel phosphorylation +p hsa04110
#> # ℹ 154 more rows
#> # ℹ 28 more variables: from_nd , to_nd ,
#> # SRR14509882 , SRR14509883 ,
#> # SRR14509884 , SRR14509885 ,
#> # SRR14509886 , SRR14509887 ,
#> # SRR14509888 , SRR14509889 ,
#> # SRR14509890 , SRR14509891 , …边值
相同的效果可以通过 获得,使用命名数值向量作为输入。此函数根据节点值添加边值。以下示例显示了将 LFC 组合到边缘。这与 的行为不同。edge_matrixedge_numeric_sumedge_numeric
## Numeric vector (name is SYMBOL)
vinflfc setNames(row.names(vinf))
g |>
## Use graphics_name to merge
mutate(grname=strsplit(graphics_name, ",") |> vapply("[", 1, FUN.VALUE="a")) |>
activate(edges) |>
mutate(summed = edge_numeric_sum(vinflfc, name="grname")) |>
filter(!is.na(summed)) |>
activate(nodes) |>
mutate(x=NULL, y=NULL, deg=centrality_degree(mode="all")) |>
filter(deg>0) |>
ggraph(layout="nicely")+
geom_edge_parallel(aes(color=summed, width=summed,
linetype=subtype_name),
arrow=arrow(length=unit(1,"mm")),
start_cap=circle(2,"mm"),
end_cap=circle(2,"mm"))+
geom_node_point(aes(fill=I(bgcolor)))+
geom_node_text(aes(label=grname,
filter=type=="gene"),
repel=TRUE, bg.colour="white")+
scale_edge_width(range=c(0.1,2))+
scale_edge_color_gradient(low="blue", high="red", name="Edge")+
theme_void()
可视化多重富集结果
您可以可视化多个富集分析的结果。与将函数与类一起使用类似,可以在函数中使用一个函数。通过向此功能提供对象,如果结果中存在可视化的通路,则通路内的基因信息可以反映在图中。在这个例子中,除了上面提到的尿路上皮细胞的变化外,还比较了肾近端肾小管上皮细胞的变化(Assetta等人,2016)。ggkeggenrichResultappend_cpmutateenrichResult
## These are RDAs storing DEGs
load("degListRPTEC.rda")
load("degURO.rda")
library(org.Hs.eg.db);
library(clusterProfiler);
input_uro mutate(uro=append_cp(ekuro, how="all"),
rptec=append_cp(ekrptec, how="all"),
converted_name=convert_id("hsa"))
ggraph(g1, layout="manual", x=x, y=y) +
geom_edge_parallel(width=0.5, arrow = arrow(length = unit(1, 'mm')),
start_cap = square(1, 'cm'),
end_cap = square(1.5, 'cm'), aes(color=subtype_name))+
geom_node_rect(aes(fill=uro, xmax=x, filter=type=="gene"))+
geom_node_rect(aes(fill=rptec, xmin=x, filter=type=="ge服务器托管网ne"))+
scale_fill_manual(values=c("steelblue","tomato"), name="urothelial|rptec")+
ggfx::with_outer_glow(geom_node_text(aes(label=converted_name, filter=type!="group"), size=2), colour="white", expand=1)+
theme_void()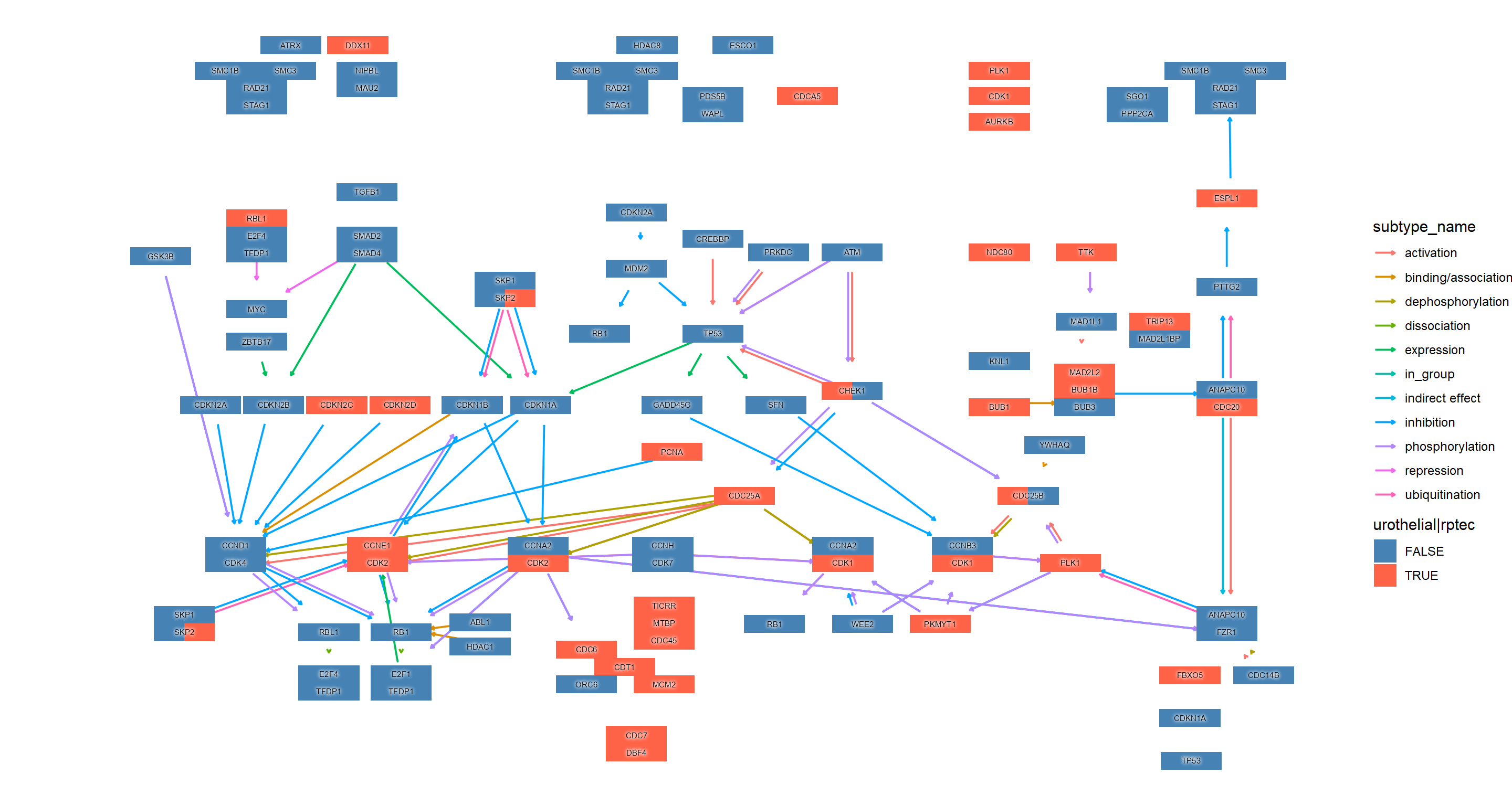
我们可以按 组合多个图。rawMappatchwork
library(patchwork)
comb 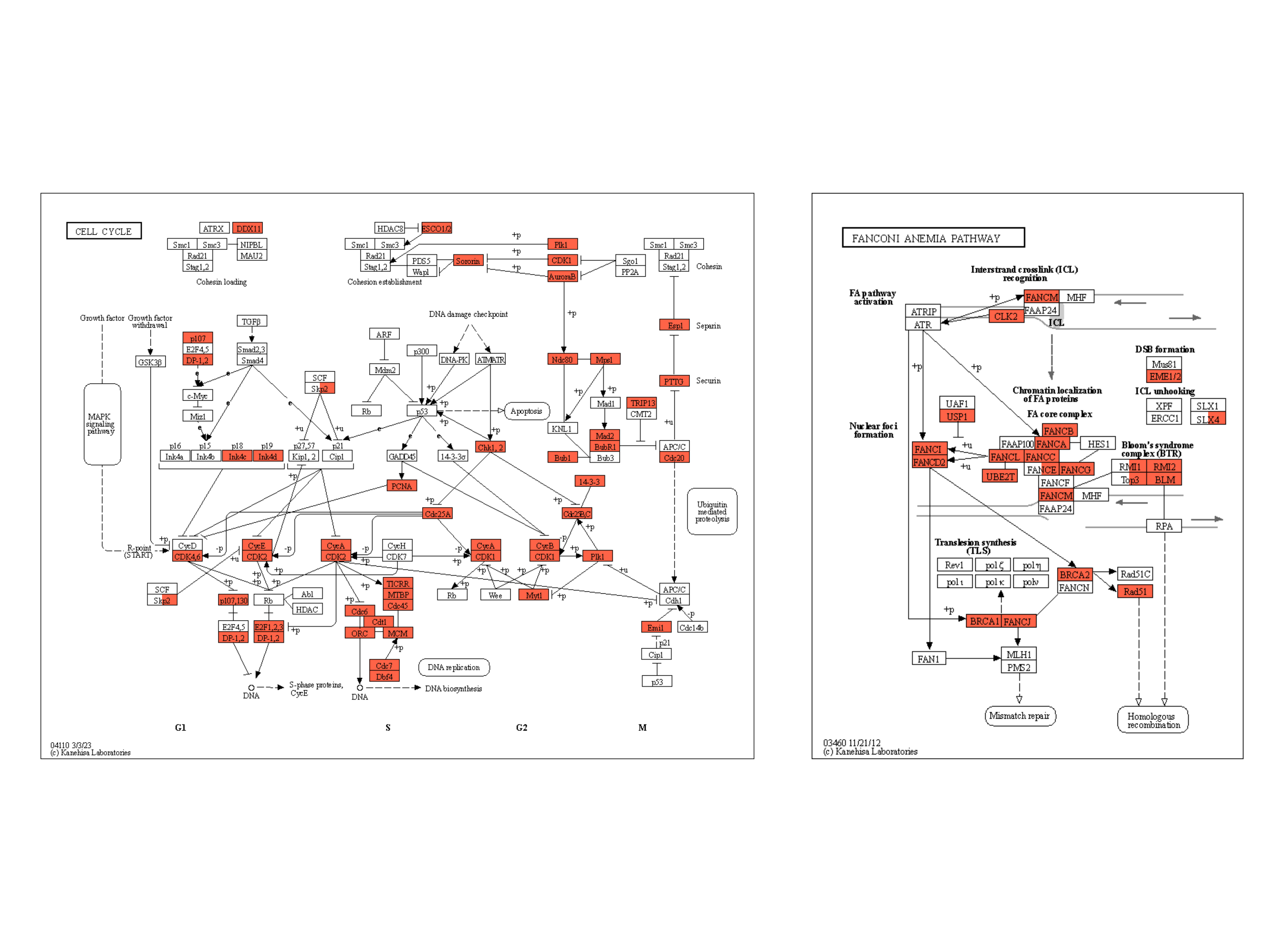
下面的示例将类似的反射应用于原始 KEGG 图谱,并突出显示在两种条件下都显示出统计学显着变化的基因,使用黄色外光,由 clusterProfiler 生成的组成,富集结果为 。ggfxdotplotpatchwork
right
mutate(uro=append_cp(ekuro, how="all"),
rptec=append_cp(ekrptec, how="all"),
converted_name=convert_id("hsa"))
gg 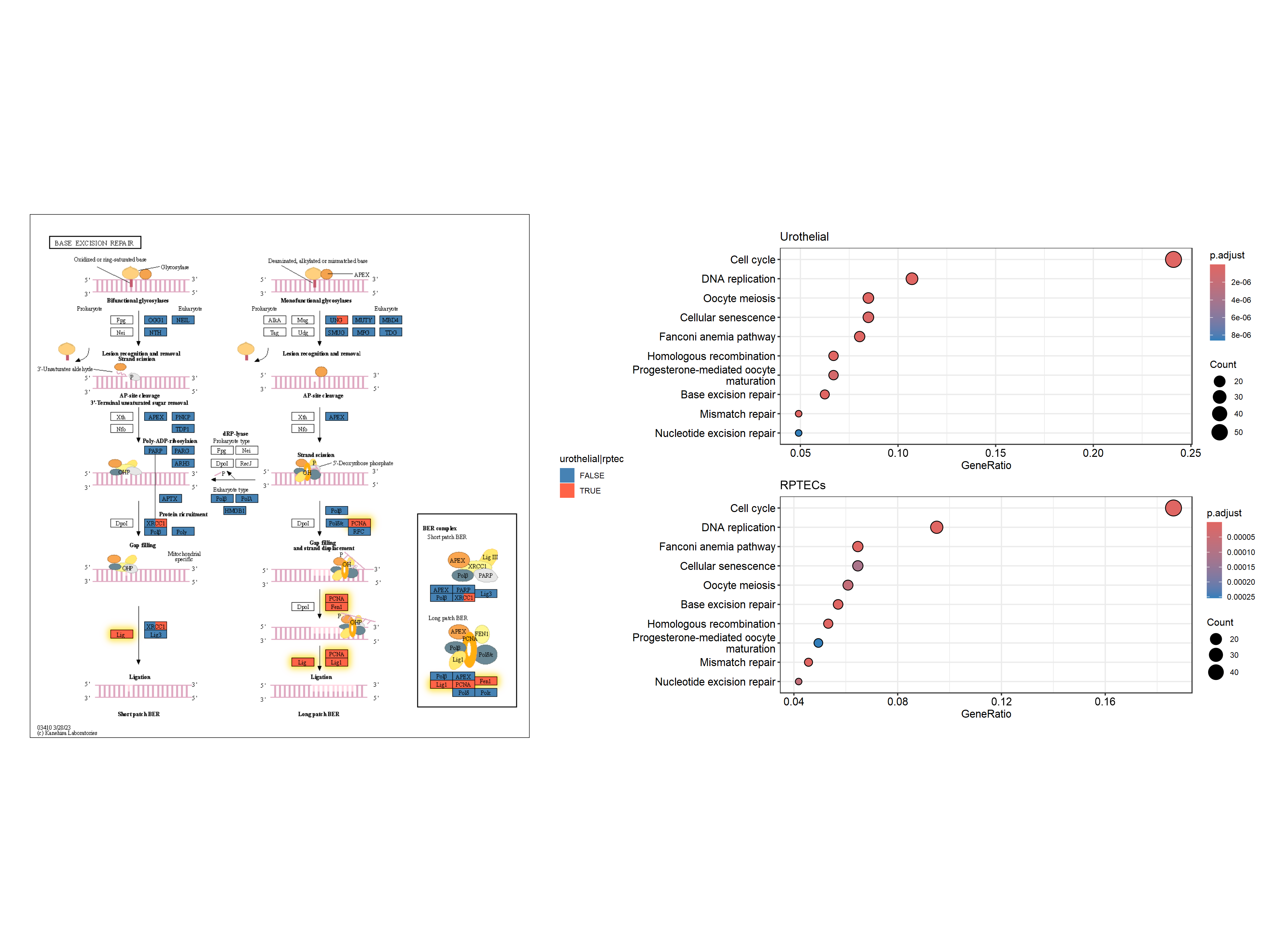
跨多个通路的多重富集分析结果
除了天然布局外,有时还可以在多个通路中显示有趣的基因,例如DEGs。在这里,我们使用散点图库来可视化跨多个途径的多个富集分析结果。
library(scatterpie)
## Obtain enrichment analysis results
entrezid
clusterProfiler::bitr("SYMBOL","ENTREZID",org.Hs.eg.db)
cp
clusterProfiler::bitr("SYMBOL","ENTREZID",org.Hs.eg.db)
cp2 row.names())[c(1,3,4)]
pathways [1] "hsa04110" "hsa03460" "hsa03440"我们获得多个通路数据(该函数返回原生坐标,但我们忽略它们)。
g1 mutate(new_name=
ifelse(name=="undefined",
paste0(name,"_",pathway_id,"_",orig.id),
name)) |>
convert(to_contracted, new_name, simplify=FALSE) |>
activate(nodes) |>
mutate(purrr::map_vec(.orig_data,function (x) x[1,] )) |>
mutate(pid1 = purrr::map(.orig_data,function (x) unique(x["pathway_id"]) )) |>
mutate(hsa03440 = purrr:::map_lgl(pid1, function(x) "hsa03440" %in% x$pathway_id) ,
hsa04110 = purrr:::map_lgl(pid1, function(x) "hsa04110" %in% x$pathway_id),
hsa03460 = purrr:::map_lgl(pid1, function(x) "hsa03460" %in% x$pathway_id))
nds activate(nodes) |> data.frame()
eds activate(edges) |> data.frame()
rmdup_eds activate(nodes) |>
mutate(
in_pathway_uro=append_cp(cp, pid=include,name="new_name"),
x=NULL, y=NULL,
in_pathway_rptec=append_cp(cp2, pid=include,name = "new_name"),
id=convert_id("hsa",name = "new_name")) |>
morph(to_subgraph, type!="group") |>
mutate(deg=centrality_degree(mode="all")) |>
unmorph() |>
filter(deg>0)在这里,我们还将基于图的聚类结果分配给图,并缩放节点的大小,以便节点可以通过散点图可视化。
V(g2_2)$walktrap 最后,我们用于可视化。背景散点表示基因是否在通路中,前景表示基因是否在多个数据集中差异表达。我们突出显示了在两个数据集中通过金色差异表达的基因。geom_scatterpie
g4 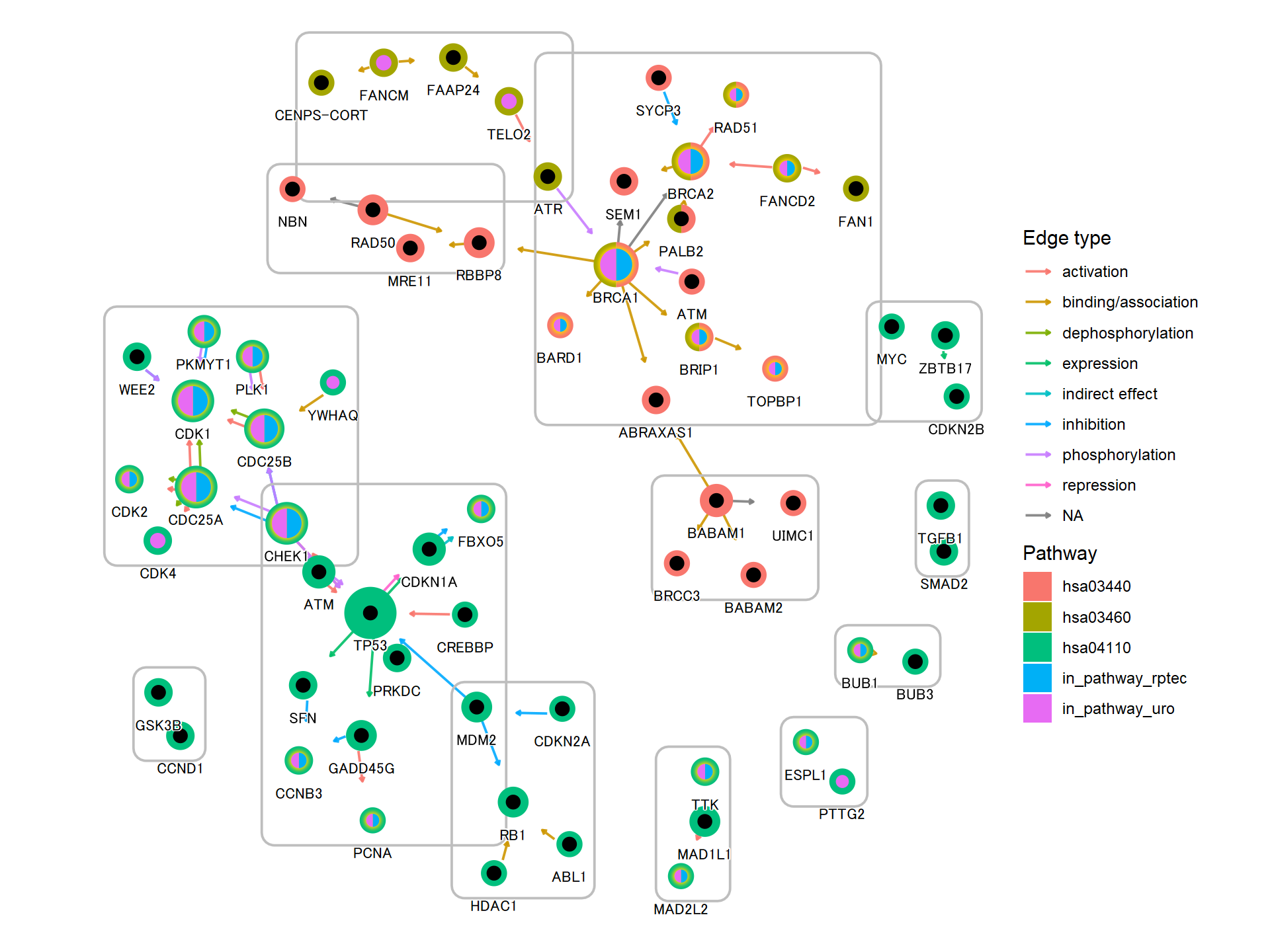
5.4在KEGG图谱上投影基因调控网络
使用此软件包,可以将推断的网络(例如基因调控网络或由其他软件推断的 KO 网络)投射到 KEGG 图谱上。以下是使用 将 CBNplot 推断的通路内的 KO 网络子集投影到相应通路的参考图上的示例。当然,也可以投影使用其他方法创建的网络。MicrobiomeProfiler
library(dplyr)
library(igraph)
library(tidygraph)
library(CBNplot)
library(ggkegg)
library(MicrobiomeProfiler)
data(Rat_data)
ko.res % magrittr::set_rownames(value=Rat_data) %>% magrittr::set_colnames(value=paste0('S', seq_len(ncol(.))))
returnnet 绘制生成的地图。在此示例中,估计的强度首先用彩色边缘显示,然后参考图的边缘在其顶部以黑色绘制。此外,两个图形中包含的边缘都以黄色突出显示。CBNplot
## Summarize duplicate edges including `strength` attribute
number activate(edges) |> data.frame() |> group_by(from,to) |>
summarise(n=n(), incstr=sum(!is.na(strength)))
## Annotate them
joined activate(edges) |> full_join(number) |> mutate(both=n>1&incstr>0)
joined |>
activate(nodes) |>
filter(!is.na(type)) |>
mutate(convertKO=convert_id("ko")) |>
activate(edges) |>
ggraph(x=x, y=y) +
geom_edge_link0(width=0.5,aes(filter=!is.na(strength),
color=strength), linetype=1)+
ggfx::with_outer_glow(
geom_edge_link0(width=0.5,aes(filter=!is.na(strength) & both,
color=strength), linetype=1),
colour="yellow", sigma=1, expand=1)+
geom_edge_link0(width=0.1, aes(filter=is.na(strength)))+
scale_edge_color_gradient(low="blue",high="red")+
geom_node_rect(color="black", aes(fill=type))+
geom_node_text(aes(label=convertKO), size=2)+
geom_node_text(aes(label=ifelse(grepl(":", graphics_name), strsplit(graphics_name, ":") |>
sapply("[",2) |> stringr::str_wrap(22), stringr::str_wrap(graphics_name, 22)),
filter=!is.na(type) & type=="map"), family="serif",
size=2, na.rm=TRUE)+
theme_void()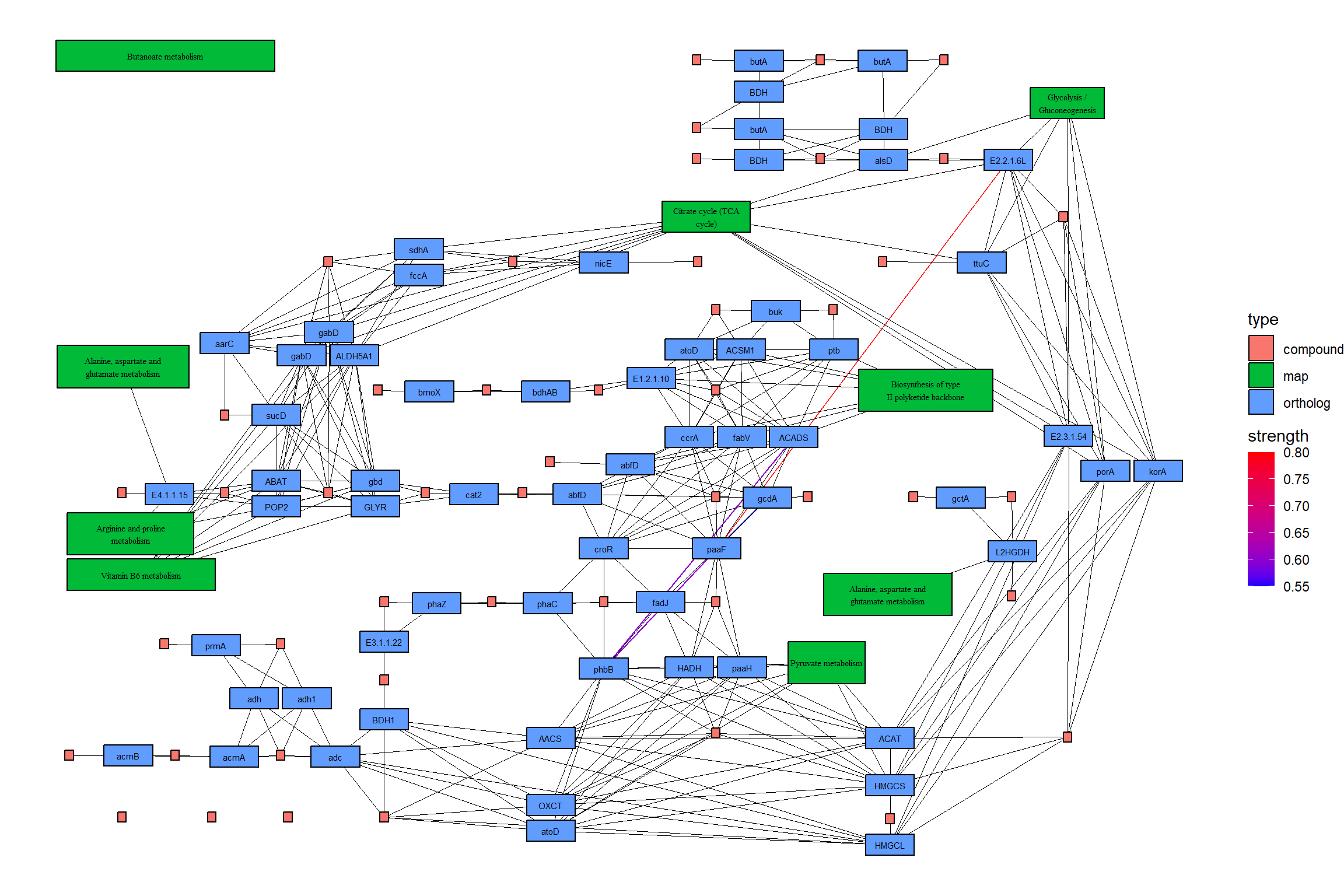
5.4.1投影到原始 KEGG 地图上
您可以直接将推断网络投影到原始 PATHWAY 地图上,这样可以直接比较您自己的数据集中精选数据库和推断网络的知识。
raws
ggraph(x=x, y=y) +
geom_edge_link(width=0.5,aes(filter=!is.na(strength),
color=strength),
linetype=1,
arrow=arrow(length=unit(1,"mm"),type="closed"),
end_cap=circle(5,"mm"))+
scale_edge_color_gradient2()+
overlay_raw_map(transparent_colors = c("#ffffff"))+
theme_void()
raws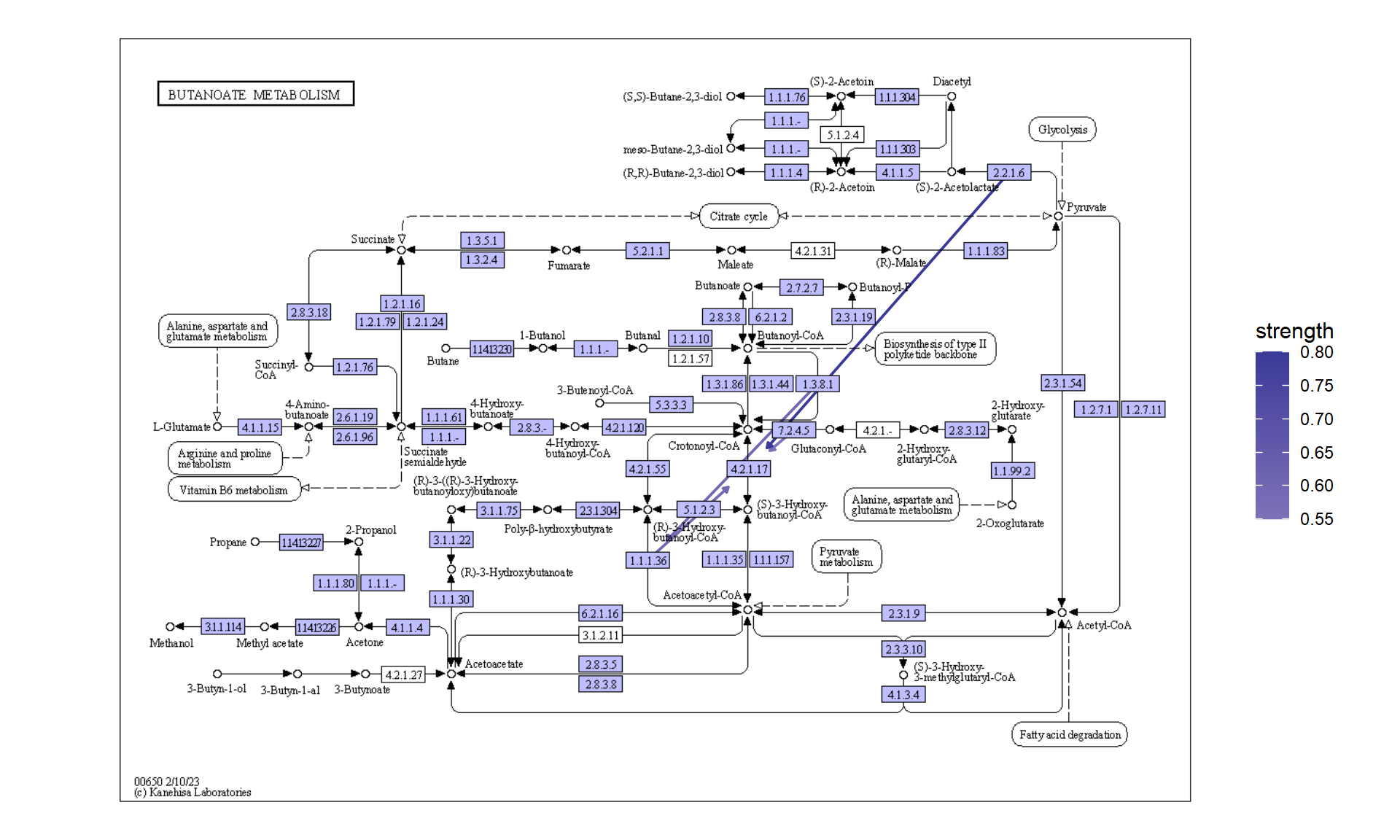
5.5分析单细胞转录组学中的簇标记基因
该软件包也可应用于单细胞分析。例如,考虑将簇之间的标记基因映射到 KEGG 通路上,并将它们与降维图一起绘制。在这里,我们使用包。我们进行基本面分析。Seurat
library(Seurat)
library(dplyr)
# dir = "../filtered_gene_bc_matrices/hg19"
# pbmc.data 随后,我们绘制了PCA降维的结果。
其中,在本研究中,我们对簇 1 和 5 的标记基因进行了富集分析。
library(clusterProfiler)
## Directly access slots in Seurat
pcas
`colnames% group_by(Cell) %>%
mutate(meanX=mean(PC_1), meanY=mean(PC_2))) |>
select(Cell, meanX, meanY)
label filter(cluster=="1" & p_val_adj
dplyr::select(gene))$gene,fromType="SYMBOL",toType="ENTREZID",OrgDb = org.Hs.eg.db)$ENTREZID
marker_5 filter(cluster=="5" & p_val_adj
dplyr::select(gene))$gene,fromType="SYMBOL",toType="ENTREZID",OrgDb = org.Hs.eg.db)$ENTREZID
mk1_enrich 从中获取颜色信息,并使用 获取通路。在这里,我们选择了 ,节点根据降维图中的颜色着色,两个聚类中的标记都按指定的颜色 () 着色。这促进了通路信息(如KEGG)与单细胞分析数据之间的联系,从而能够创建直观且易于理解的视觉表示。ggplot2ggkeggOsteoclast differentiation (hsa04380)ggfxtomato
## Make color map
built mutate(marker_1=append_cp(mk1_enrich),
marker_5=append_cp(mk5_enrich))
gg 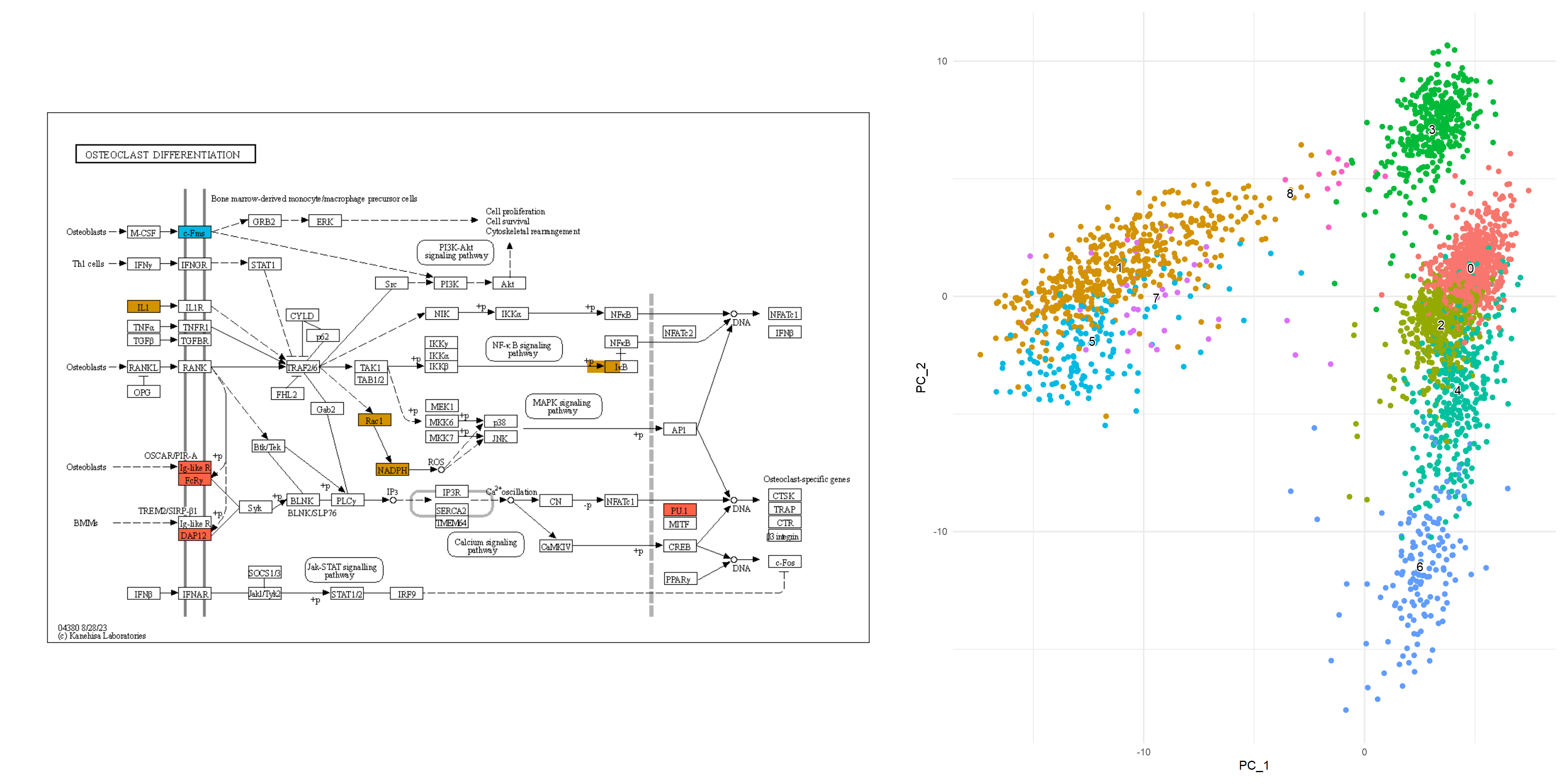
5.5.1组成多个通路的示例
我们可以在多种途径中检查标记基因,以更好地了解标记基因的作用。
library(clusterProfiler)
library(org.Hs.eg.db)
subset_lab filter(cluster=="1" & p_val_adj
dplyr::select(gene))$gene,fromType="SYMBOL",toType="ENTREZID",OrgDb = org.Hs.eg.db)$ENTREZID
marker_5 filter(cluster=="5" & p_val_adj
dplyr::select(gene))$gene,fromType="SYMBOL",toType="ENTREZID",OrgDb = org.Hs.eg.db)$ENTREZID
marker_6 filter(cluster=="6" & p_val_adj
dplyr::select(gene))$gene,fromType="SYMBOL",toType="ENTREZID",OrgDb = org.Hs.eg.db)$ENTREZID
marker_4 filter(cluster=="4" & p_val_adj
dplyr::select(gene))$gene,fromType="SYMBOL",toType="ENTREZID",OrgDb = org.Hs.eg.db)$ENTREZID
mk1_enrich mutate(marker_4=append_cp(mk4_enrich),
marker_6=append_cp(mk6_enrich),
gene_name=convert_id("hsa"))
gg1 mutate(marker_1=append_cp(mk1_enrich),
marker_5=append_cp(mk5_enrich))
gg2 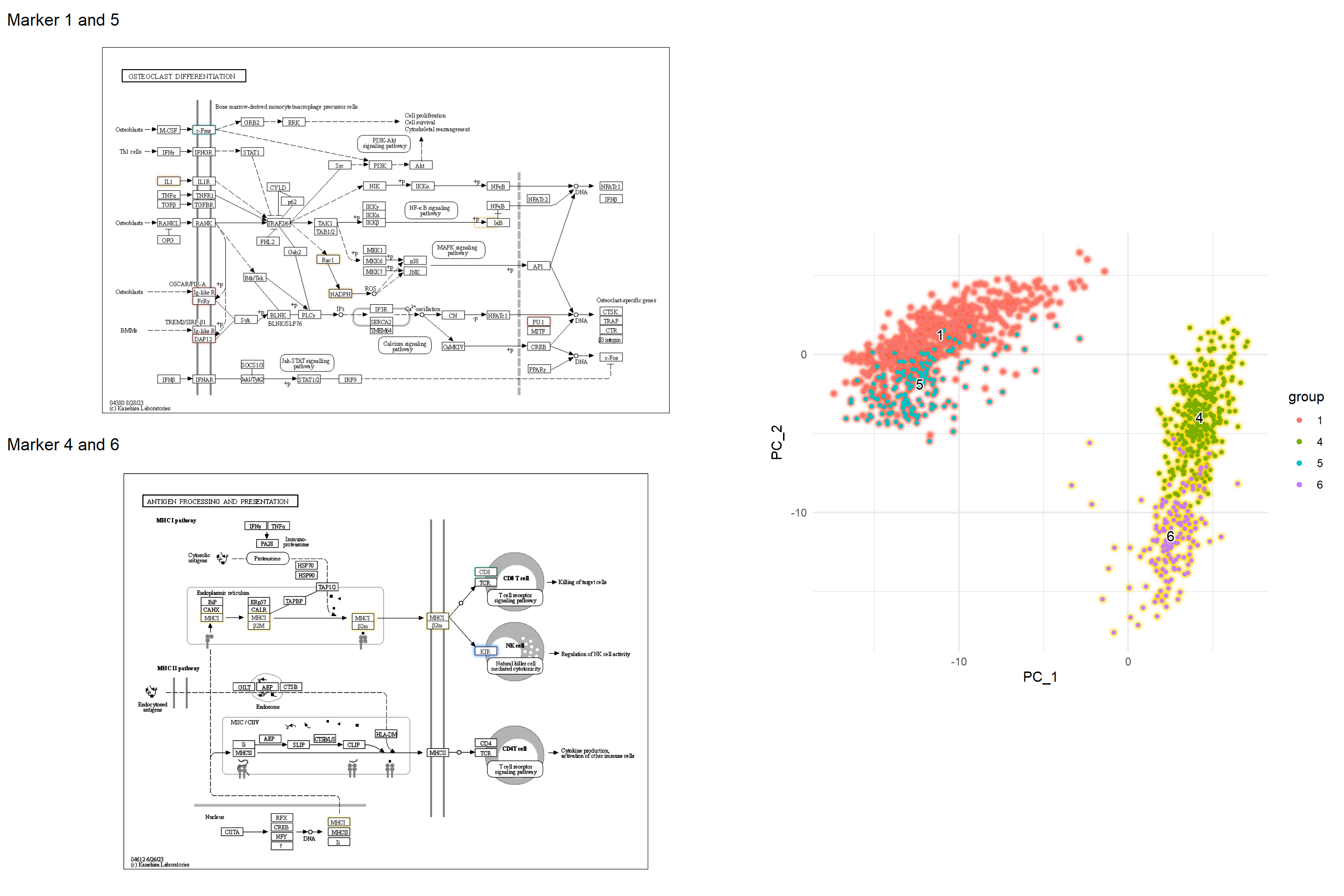
5.5.2原始地图上数值的条形图
对于它们在多个聚类中丰富的节点,我们可以绘制数值的条形图。引用的代码由 inscaven提供。
## Assign lfc to graph
mark_4 filter(cluster=="4" & p_val_adj
dplyr::select(gene))$gene,fromType="SYMBOL",toType="ENTREZID",OrgDb = org.Hs.eg.db)
mark_6 filter(cluster=="6" & p_val_adj
dplyr::select(gene))$gene,fromType="SYMBOL",toType="ENTREZID",OrgDb = org.Hs.eg.db)
mark_4$lfc mutate(mk4lfc=node_numeric(mk4lfc),
mk6lfc=node_numeric(mk6lfc))
## Make data frame containing necessary data from node
subset_df activate(nodes) |> data.frame() |>
dplyr::filter(marker_4 & marker_6) |>
dplyr::select(orig.id, mk4lfc, mk6lfc, x, y, xmin, xmax, ymin, ymax) |>
tidyr::pivot_longer(cols=c("mk4lfc","mk6lfc"))
## Actually we dont need position list
pos_list unique()) {
tmp unique()
ymax unique()
xmin unique()
xmax unique()
pos_list[[as.character(i)]]
ggplot(aes(x=name, y=value, fill=name))+
geom_col(width=1)+
scale_fill_manual(values=c(gr_cols["4"] |> as.character(),
gr_cols["6"] |> as.character()))+
labs(x = NULL, y = NULL) +
coord_cartesian(expand = FALSE) +
theme(
legend.position = "none",
panel.background = element_rect(fill = "transparent", colour = NA),
line = element_blank(),
text = element_blank()
)
gbar 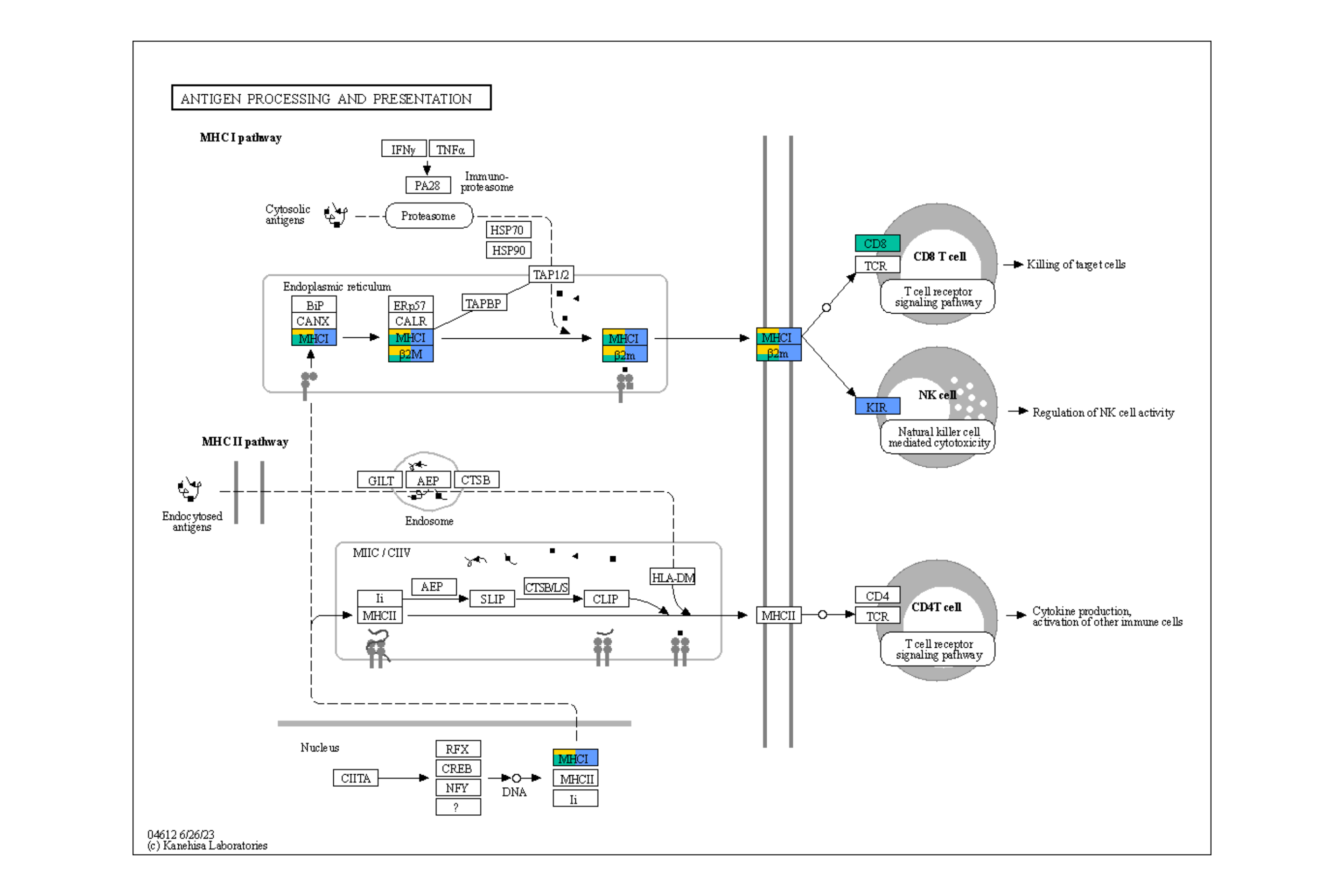
5.5.3所有聚类的条形图
通过迭代上述代码,我们可以将所有聚类的定量数据绘制在图上。虽然最好使用 ggplot2 映射来生成图例,但这里我们从降维图中获取图例。
g1 filter(cluster==cluster_num & p_val_adj
dplyr::select(gene))$gene,fromType="SYMBOL",toType="ENTREZID",OrgDb = org.Hs.eg.db)
mark$lfc mutate(!!coln := node_numeric(mark$lfc |> setNames(mark$hsa)))
}做。ggplotGrob()
subset_df activate(nodes) |> data.frame() |>
dplyr::select(orig.id, paste0("marker",seq_len(9)-1,"lfc"), x, y, xmin, xmax, ymin, ymax) |>
tidyr::pivot_longer(cols=paste0("marker",seq_len(9)-1,"lfc"))
pos_list unique()) {
tmp unique()
ymax unique()
xmin unique()
xmax unique()
pos_list[[as.character(i)]] filter(!is.na(value)) |> dim())[1]!=0) {
barp filter(!is.na(value)) |>
ggplot(aes(x=name, y=value, fill=name))+
geom_col(width=1)+
scale_fill_manual(values=all_gr_cols)+
## We add horizontal line to show the direction of bar
geom_hline(yintercept=0, linewidth=1, colour="grey")+
labs(x = NULL, y = NULL) +
coord_cartesian(expand = FALSE) +
theme(
legend.position = "none",
panel.background = element_rect(fill = "transparent", colour = NA),
text = element_blank()
)
gbar 获取图例并进行修改。
## Take scplot legend, make it rectangle
## Make pseudo plot
dd2 
## Make dummy legend by `fill`
graph_tmp setNames("transparent"))+
theme_void()
## Overlaid the raw map
overlaid 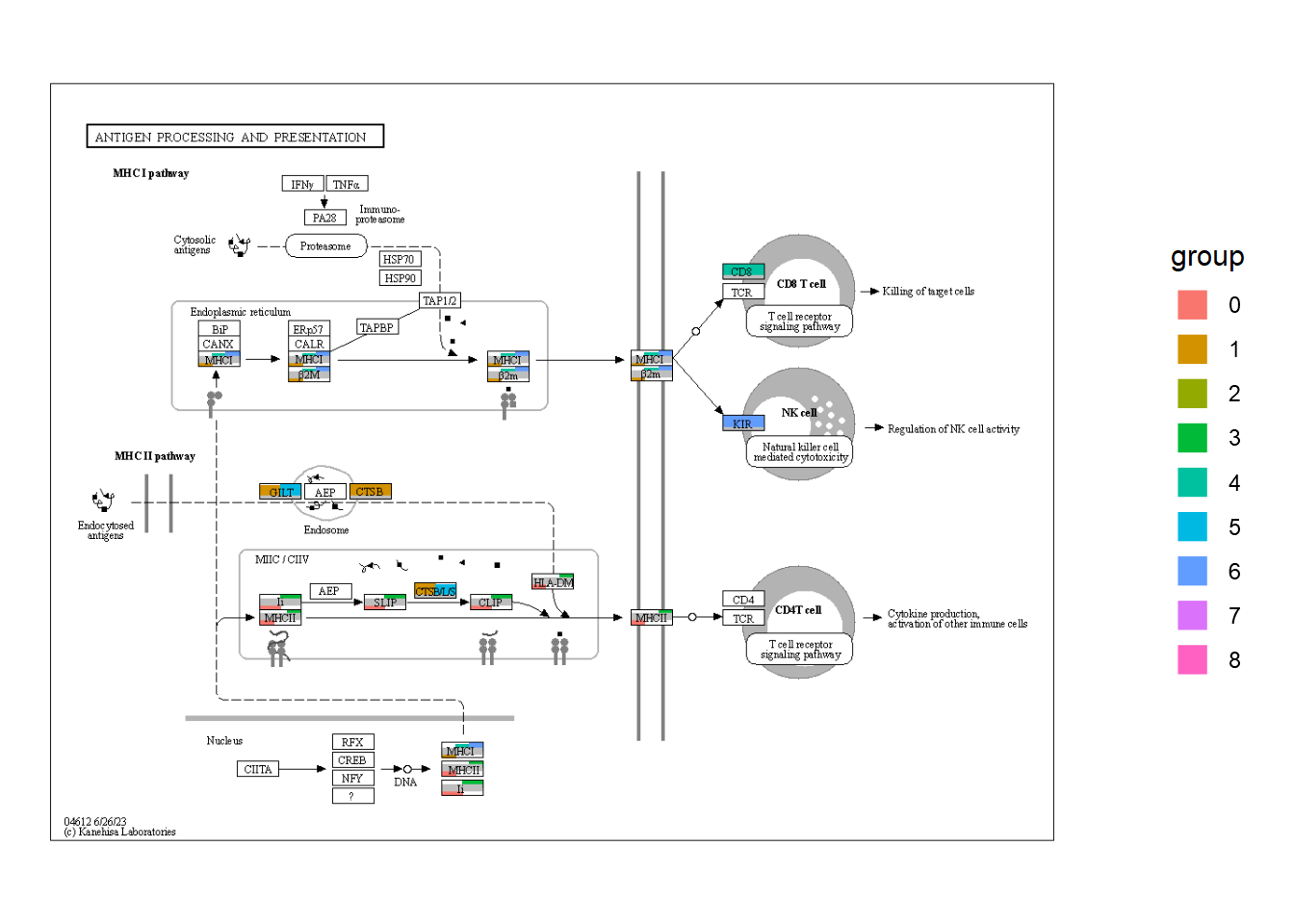
5.6自定义全局地图可视化
使用的一个优点是利用 和 的强大功能有效地可视化全球地图。在这里,我展示了一个可视化从全球地图中的一些微生物组实验中获得的 log2 倍数变化值的示例。首先,我们加载必要的数据,这些数据可以从调查 KO 的数据集中获得,这些数据是从管道中获得的,例如 .ggkeggggplot2ggraphHUMAnN3
load("../lfcs.rda") ## Storing named vector of KOs storing LFCs and significant KOs
load("../func_cat.rda") ## Functional categories for hex values in ko01100
lfcs |> head()
#> ko:K00013 ko:K00018 ko:K00031 ko:K00042 ko:K00065
#> -0.2955686 -0.4803597 -0.3052872 0.9327130 1.0954976
#> ko:K00087
#> 0.8713860
signame |> head()
#> [1] "ko:K00013" "ko:K00018" "ko:K00031" "ko:K00042"
#> [5] "ko:K00065" "ko:K00087"
func_cat |> head()
#> # A tibble: 6 3
#> hex class top
#>
#> 1 #B3B3E6 Metabolism; Carbohydrate metabolism Amin…
#> 2 #F06292 Metabolism; Biosynthesis of other secondary… Bios…
#> 3 #FFB3CC Metabolism; Metabolism of cofactors and vit… Bios…
#> 4 #FF8080 Metabolism; Nucleotide metabolism Puri…
#> 5 #6C63F6 Metabolism; Carbohydrate metabolism Glyc…
#> 6 #FFCC66 Metabolism; Amino acid metabolism Bios…
## Named vector for Assigning functional category
hex setNames(func_cat$hex)
class setNames(func_cat$hex)
hex |> head()
#> #B3B3E6 #F06292 #FFB3CC #FF8080 #6C63F6 #FFCC66
#> "#B3B3E6" "#F06292" "#FFB3CC" "#FF8080" "#6C63F6" "#FFCC66"
class |> head()
#> #B3B3E6
#> "Metabolism; Carbohydrate metabolism"
#> #F06292
#> "Metabolism; Biosynthesis of other secondary metabolites"
#> #FFB3CC
#> "Metabolism; Metabolism of cofactors and vitamins"
#> #FF8080
#> "Metabolism; Nucleotide metabolism"
#> #6C63F6
#> "Metabolism; Carbohydrate metabolism"
#> #FFCC66
#> "Metabolism; Amino acid metabolism"预处理
我们得到了 ko01100,并处理了图形。首先,我们附加与化合物间关系相对应的边。尽管大多数反应是可逆的,并且默认情况下会在 中添加两条边,但我们在此处指定用于可视化。此外,转换化合物 ID 和 KO ID 并将属性附加到图形中。tbl_graphprocess_reactionsingle_edge=TRUE
g process_reaction(single_edge=TRUE)
g mutate(x=NULL, y=NULL)
g activate(nodes) |> mutate(compn=convert_id("compound",
first_arg_comma = FALSE))
g activate(edges) |> mutate(kon=convert_id("ko",edge=TRUE))接下来,我们将 KO 和度数等值附加到图表中。此外,在这里,我们将其他属性(例如哪些物种具有酶)附加到图表中。此类信息可以从 的分层输出中获得。HUMAnN3
g2 activate(edges) |>
mutate(kolfc=edge_numeric(lfcs), ## Pre-computed LFCs
siglgl=.data$name %in% signame) |> ## Whether the KO is significant
activate(nodes) |>
filter(type=="compound") |> ## Subset to compound nodes and
mutate(Degree=centrality_degree(mode="all")) |> ## Calculate degree
activate(nodes) |>
filter(Degree>2) |> ## Filter based on degree
activate(edges) |>
mutate(Species=ifelse(kon=="lyxK", "Escherichia coli", "Others"))接下来,我们根据 ko01100 检查这些 KO 的总体类别,KO 数量最多的类别是碳水化合物代谢。
class_table activate(edges) |>
mutate(siglgl=name %in% signame) |>
filter(siglgl) |>
data.frame())$fgcolor |>
table() |> sort(decreasing=TRUE)
names(class_table) Metabolism; Carbohydrate metabolism
#> 20
#> Metabolism; Glycan biosynthesis and metabolism
#> 16
#> Metabolism; Metabolism of cofactors and vitamins
#> 11
#> Metabolism; Amino acid metabolism
#> 8
#> Metabolism; Nucleotide metabolism
#> 7
#> Metabolism; Metabolism of terpenoids and polyketides
#> 3
#> Metabolism; Energy metabolism
#> 3
#> Metabolism; Xenobiotics biodegradation and metabolism
#> 3
#> Metabolism; Carbohydrate metabolism
#> 2
#> Metabolism; Lipid metabolism
#> 1
#> Metabolism; Biosynthesis of other secondary metabolites
#> 1
#> Metabolism; Metabolism of other amino acids
#> 1绘图
我们首先使用 和 计算度的默认值可视化整个全球地图。ko01100
ggraph(g2, layout="fr")+
geom_edge_link0(aes(color=I(fgcolor)), width=0.1)+
geom_node_point(aes(fill=I(fgcolor), size=Degree), color="black", shape=21)+
theme_graph()
我们可以将各种几何形状应用于KEGG PATHWAY中的组件,以实现有效的可视化。在此示例中,我们突出显示了由其 LFC 着色的有效边 (KO),点大小对应于网络中的度数,并显示了有效 KO 名称的边缘标签。KO名称按属性着色。这一次,我们将其设置为 和 。ggfxSpeciesEscherichia coliOthers
ggraph(g2, layout="fr") +
geom_edge_diagonal(color="grey50", width=0.1)+ ## Base edge
ggfx::with_outer_glow(
geom_edge_diagonal(aes(color=kolfc,filter=siglgl),
angle_calc = "along",
label_size=2.5),
colour="gold", expand=3
)+ ## Highlight significant edges
scale_edge_color_gradient2(midpoint = 0, mid = "white",
low=scales::muted("blue"),
high=scales::muted("red"),
name="LFC")+ ## Set gradient color
geom_node_point(aes(fill=bgcolor,size=Degree),
shape=21,
color="black")+ ## Node size set to degree
scale_size(range=c(1,4))+
geom_edge_label_diagonal(aes(
label=kon,
label_colour=Species,
filter=siglgl
),
angle_calc = "along",
label_size=2.5)+ ## Showing edge label, label color is Species attribute
scale_label_colour_manual(values=c("tomato","black"),
name="Species")+ ## Scale color for edge label
scale_fill_manual(values=hex,labels=class,name="Class")+ ## Show legend based on HEX
theme_graph()+
guides(fill = guide_legend(override.aes = list(size=5))) ## Change legend point size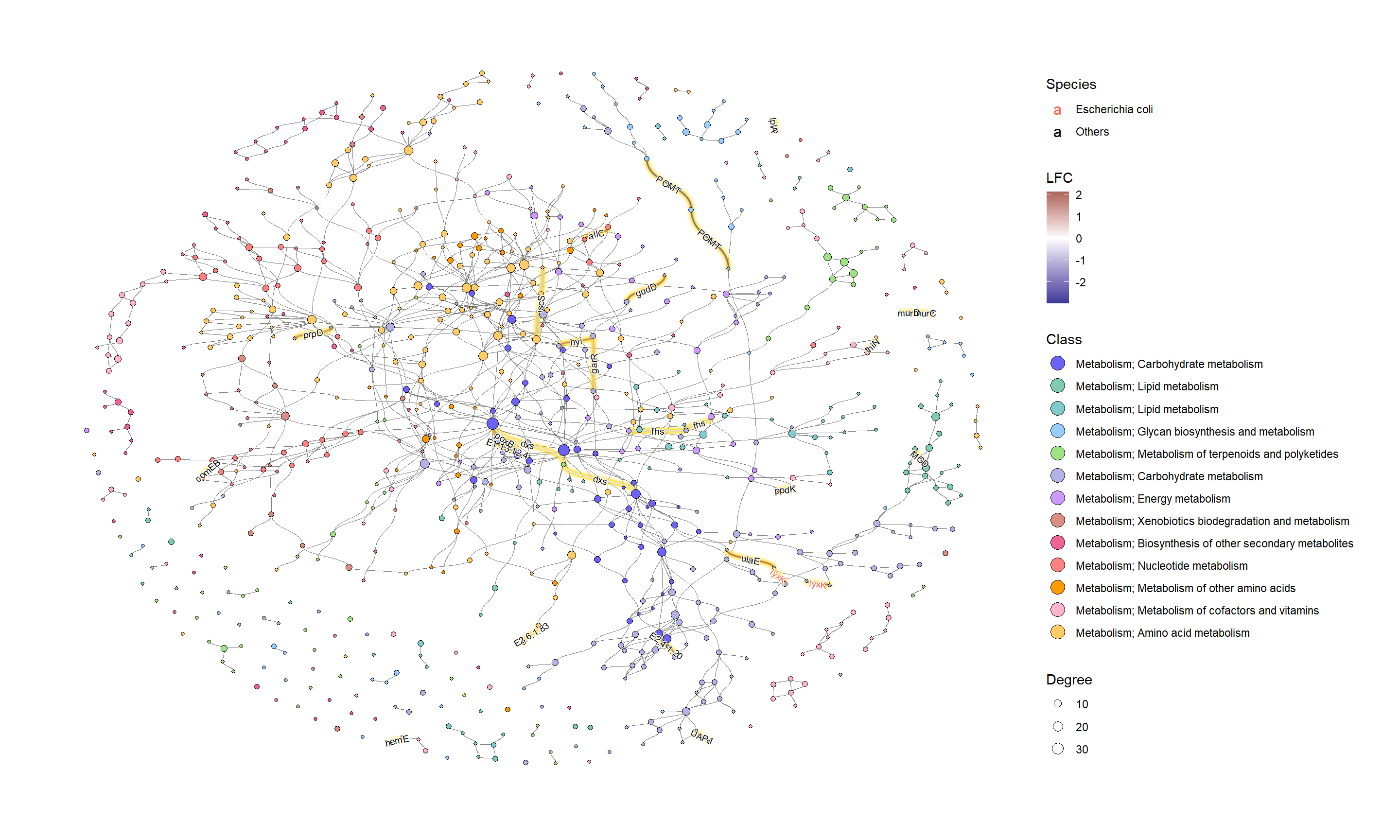
如果我们想调查特定的类,则按图中的十六进制值进行子集。
## Subset and do the same thing
g2 |>
morph(to_subgraph, siglgl) |>
activate(nodes) |>
mutate(tmp=centrality_degree(mode="all")) |>
filter(tmp>0) |>
mutate(subname=compn) |>
unmorph() |>
activate(nodes) |>
filter(bgcolor=="#B3B3E6") |>
mutate(Degree=centrality_degree(mode="all")) |> ## Calculate degree
filter(Degree>0) |>
ggraph(layout="fr") +
geom_edge_diagonal(color="grey50", width=0.1)+ ## Base edge
ggfx::with_outer_glow(
geom_edge_diagonal(aes(color=kolfc,filter=siglgl),
angle_calc = "along",
label_size=2.5),
colour="gold", expand=3
)+
scale_edge_color_gradient2(midpoint = 0, mid = "white",
low=scales::muted("blue"),
high=scales::muted("red"),
name="LFC")+
geom_node_point(aes(fill=bgcolor,size=Degree),
shape=21,
color="black")+
scale_size(range=c(1,4))+
geom_edge_label_diagonal(aes(
label=kon,
label_colour=Species,
filter=siglgl
),
angle_calc = "along",
label_size=2.5)+ ## Showing edge label
scale_label_colour_manual(values=c("tomato","black"),
name="Species")+ ## Scale color for edge label
geom_node_text(aes(label=stringr::str_wrap(subname,10,whitespace_only = FALSE)),
repel=TRUE, bg.colour="white", size=2)+
scale_fill_manual(values=hex,labels=class,name="Class")+
theme_graph()+
guides(fill = guide_legend(override.aes = list(size=5)))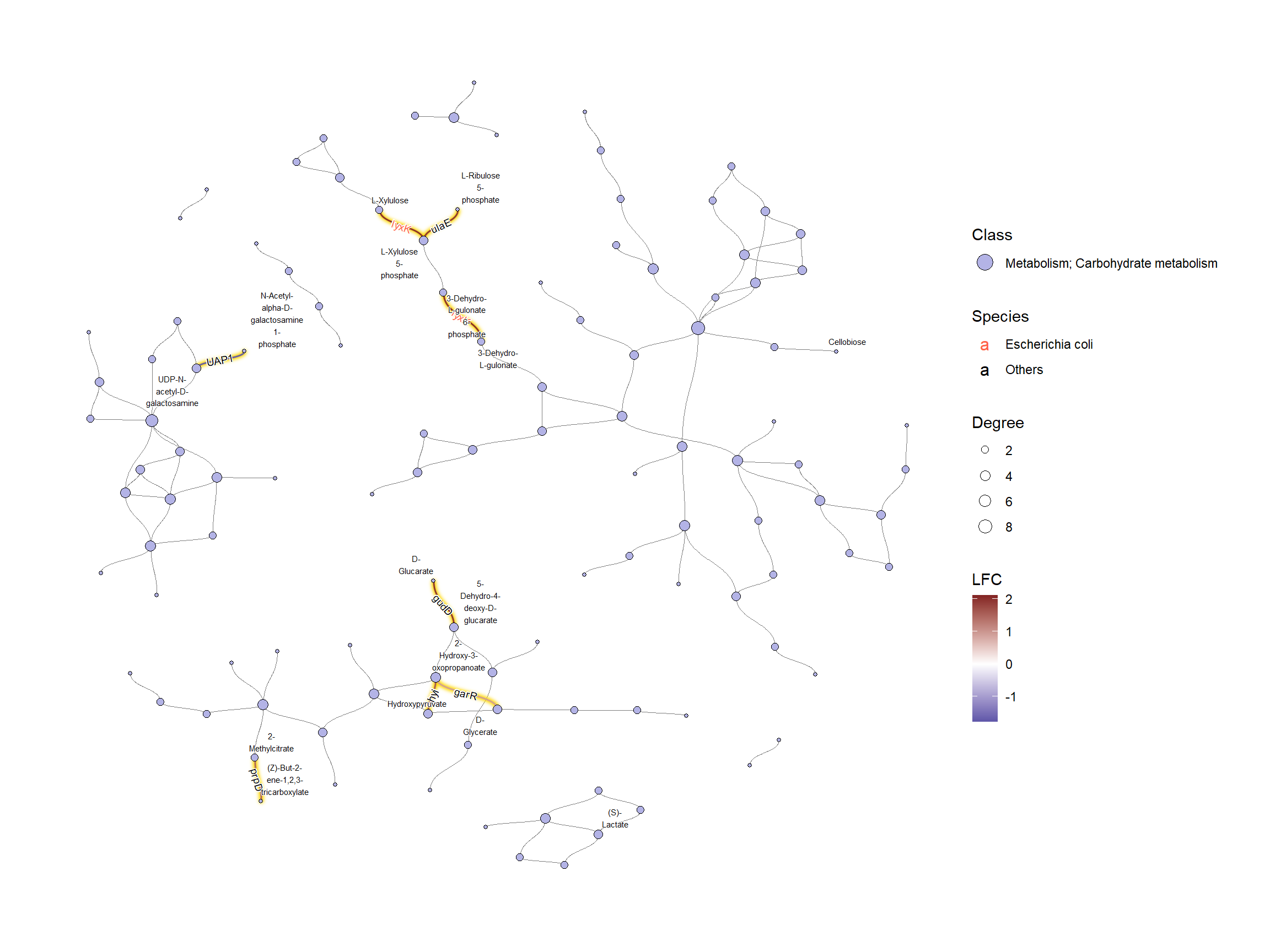
服务器托管,北京服务器托管,服务器租用 http://www.fwqtg.net
一、前言 华为宣布HarmonyOS NEXT鸿蒙星河版面向开发者开放申请,这一最新版本的鸿蒙系统也被喻为“纯血鸿蒙”。用余承东的话说便是,HarmonyOS NEXT不依赖传统的Unix内核和Linux内核,而是依靠自主的鸿蒙内核。 1月18日,在预热已久的…

