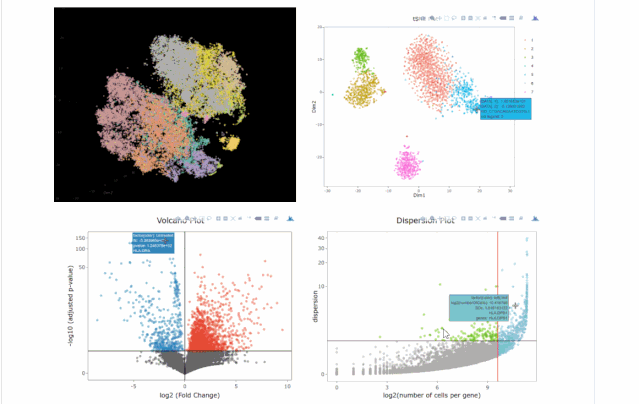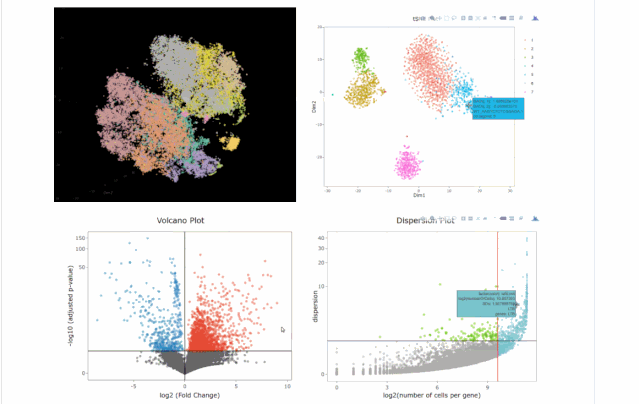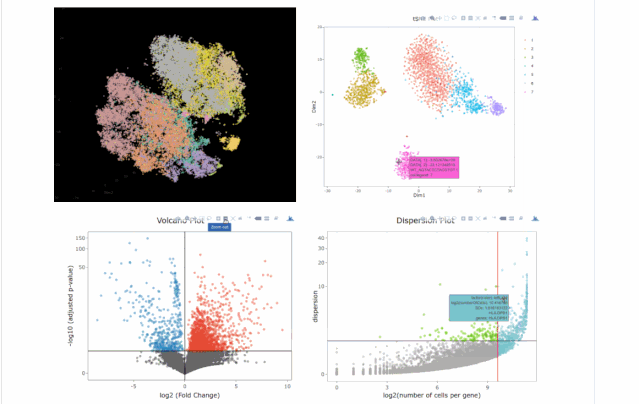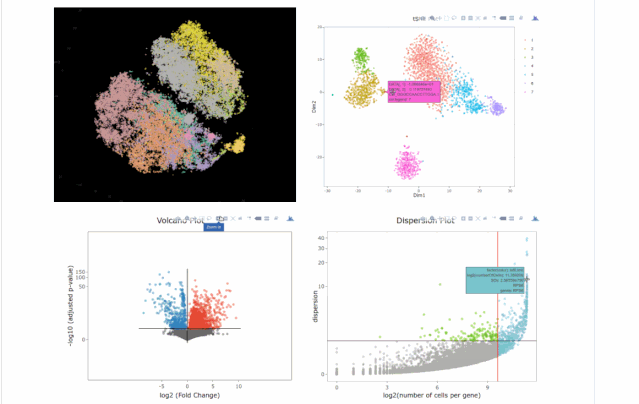
今天在翻阅single cell 的github时候,我看见了这个R包,允许我们处理各种来自单细胞测序技术的数据,如scRNA-seq,scVDJ-seq和CITE-Seq。
单细胞转录组教程汇总
想看整套的学习流程还可以戳这里:
https://vimeo.com/337822487
iCellR优点
Single(i)Cell R软件包(iCellR)在分析pipeline的每个步骤提供前所未有的灵活性,包括标准化,聚类,降维,插补,可视化等。可以通过无监督和有监督聚类进行分析, 此外,该工具包提供2D和3D交互式可视化(一个震撼的交互型3D可视化R包 – 可直接转ggplot2图为3D),差异表达分析,基于细胞、基因和聚类的filter,数据合并,dropouts的标准化,数据插补方法,校正批次差异,找到标记基因的工具聚类和条件,预测细胞类型和伪时序分析(NBT|45种单细胞轨迹推断方法比较,110个实际数据集和229个合成数据集)。感觉能做的真的好多啊!
安装iCellR
# 从镜像中进行安装
install.packages("iCellR")
# 也可以从github中进行安装
#library(devtools)
#install_github("rezakj/iCellR")
# or
#git clone https://github.com/rezakj/iCellR.git
#R
#install.packages('iCellR/', repos = NULL, type="source")下载演示数据
ion(samples = c("sample1","sample2","sample3"),# 设置需要下载的文件夹位置
setwd("/your/download/directory")
# 通过URL进行数据下载
sample.file.url = "https://s3-us-west-2.amazonaws.com/10x.files/samples/cell/pbmc3k/pbmc3k_filtered_gene_bc_matrices.tar.gz"
# 下载文件
download.file(url = sample.file.url,
destfile = "pbmc3k_filtered_gene_bc_matrices.tar.gz",
method = "auto")
# 解压缩.
untar("pbmc3k_filtered_gene_bc_matrices.tar.gz")
更多的数据可以通过这里获得:
https://genome.med.nyu.edu/results/external/iCellR/
如何使用iCellR分析scRNA-seq数据1. 加载iCellR包和下载的PBMC样本数据。library("iCellR")
my.data # 查看数据
head(my.data)[1:2]
# WT_AAACATACAACCAC-1 WT_AAACATTGAGCTAC-1
#A1BG 0 0
#A1BG.AS1 0 0
#A1CF 0 0
#A2M 0 0
#A2M.AS1 0 03. 制作iCellR类的对象。
my.obj 4. 进行质控(QC)
my.obj 5. 细胞周期预测
和上一个推送的细胞周期预测方法还是较为一致的,都是通过评分进行周期的预测,
my.obj 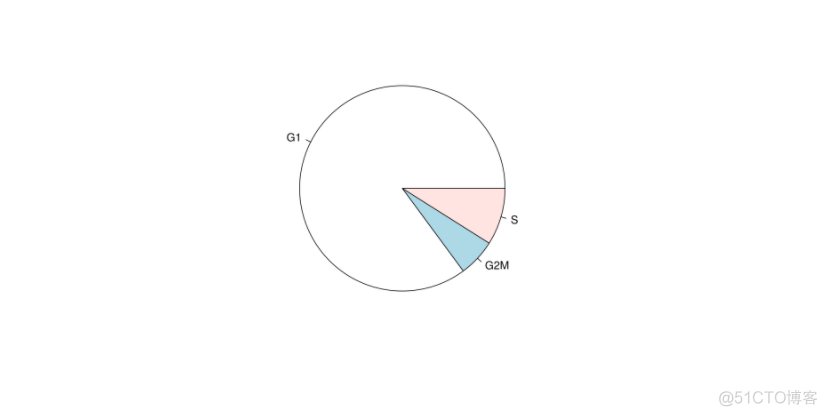
6. Plot QC
默认情况下的绘图功能都将创建交互式html文件,可以设置interactive = FALSE来关闭。
# 绘制 三个分组中UMIs, genes和 percent mito的beanplot分布图,想单独画地话可以用`?stats.plot`help一下如何修改参数
stats.plot(my.obj,
plot.type = "all.in.one",
out.name = "UMI-plot",
interactive = FALSE,
cell.color = "slategray3",
cell.size = 1,
cell.transparency = 0.5,
box.color = "red",
box.line.col = "green")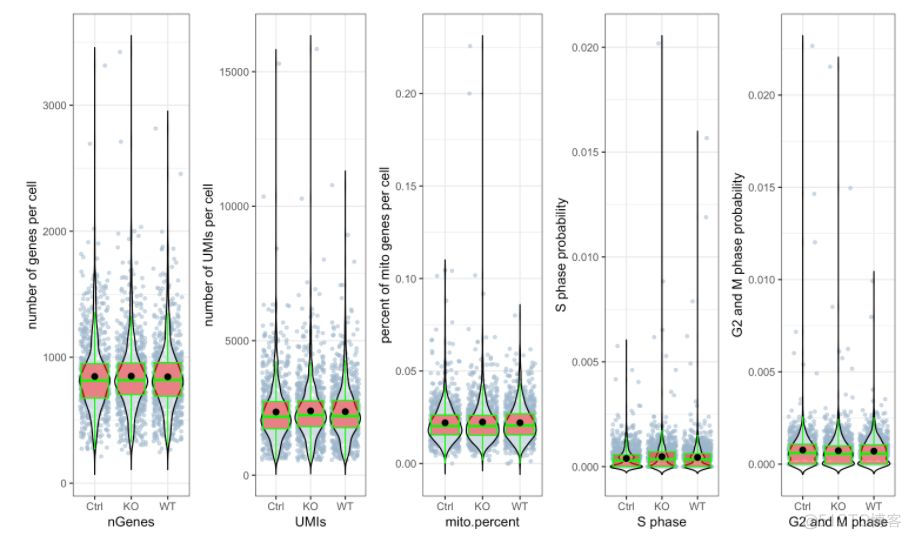
# 绘制散点图查看线粒体基因和基因分布,注意参数`plot.type`
stats.plot(my.obj, plot.type = "point.mito.umi", out.name = "mito-umi-plot")###该图为线粒体基因和UMI的相关表达分布(一般原则下会认为线粒体基因比例较高可能为破碎衰老细胞,可以设置cutoff值去掉,但如果研究为高代谢或本身耗能较多的细胞时需要考虑)
stats.plot(my.obj, plot.type = "point.gene.umi", out.name = "gene-umi-plot")###此图为UMI和GENE的表达分布(原则上遵循线性分布)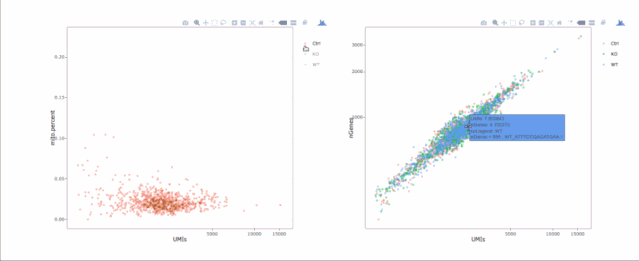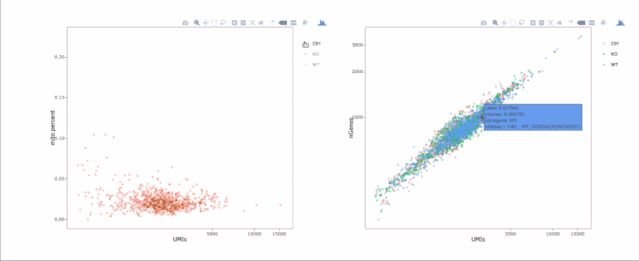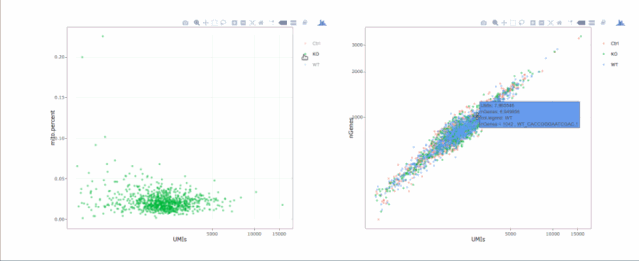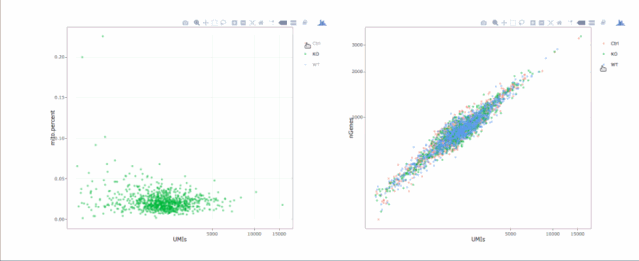
R语言 – 散点图绘制
7. 过滤细胞
my.obj # 查看剩下的细胞数(线粒体基因比例在0到0.05,基因数在200到2400之间)
dim(my.obj@main.data)
#[1] 32738 2637
# 过滤掉了63个细胞- Hemberg-lab单细胞转录组数据分析(九)- Scater包单细胞过滤
- Hemberg-lab单细胞转录组数据分析(十)- Scater基因评估和过滤
8. 进行标准化
可以根据自己的研究对数据进行标准化。还可以使用iCellR以外的工具对数据进行标准化,并将数据导入iCellR。对于大多数单细胞研究,建议使用“ranked.glsf”标准化。这种标准化化比较适合具有大量零的矩阵。且在合并数据(操作参考步骤2:汇总数据)的情况下,也能对批次进行校正。
my.obj 9. 进行二次QC
my.obj 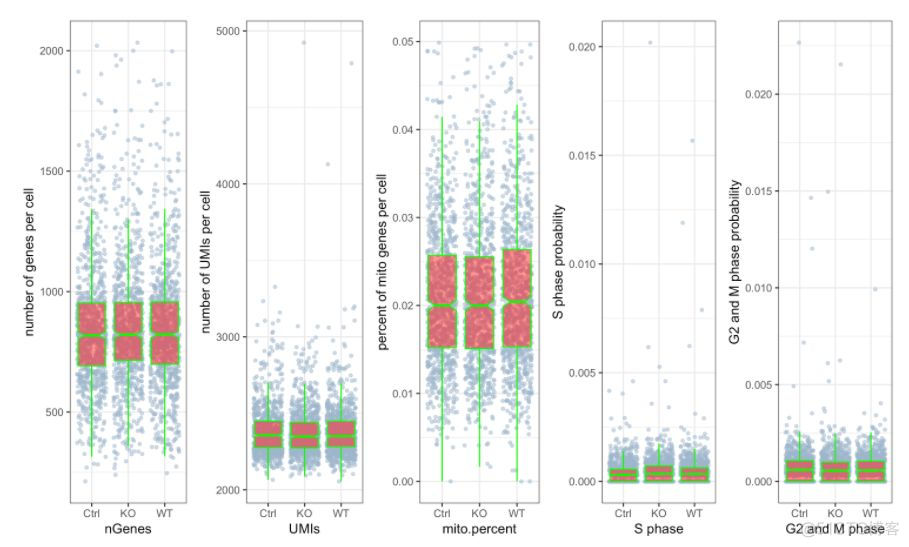
R语言 – 箱线图(小提琴图、抖动图、区域散点图)
10. Scale data
my.obj 11.进行gene统计
my.obj 12.制作聚类的基因模型
在bulk RNA-seq数据中,用平均表达值meanExp排名前500的基因(设置参数base.mean.rank对样本进行聚类是非常常见的,主要原因是为了降低噪声。在scRNA-seq数据中也可以这样做。并加上ranked.glsf归一化,会较为擅长处理具有大量零的矩阵。
# 看一下高变基因集
make.gene.model(my.obj, my.out.put = "plot",
dispersion.limit = 1.5,
base.mean.rank = 500,
no.mito.model = T,
mark.mito = T,
interactive = F,
no.cell.cycle = T,
out.name = "gene.model")
# 将高变基因集注入`my.obj` 中
my.obj 
13. 进行降维
降维是为了在信息损失较少的情况下,以比原始特征数量少得多的特征(基因)来表示数据,更好地捕捉细胞之间的关系,常见降维的方法有PCA,t-SNE,umap和diffusion map。(Hemberg-lab单细胞转录组数据分析(十一)- Scater单细胞表达谱PCA可视化)
PCA分析
my.obj 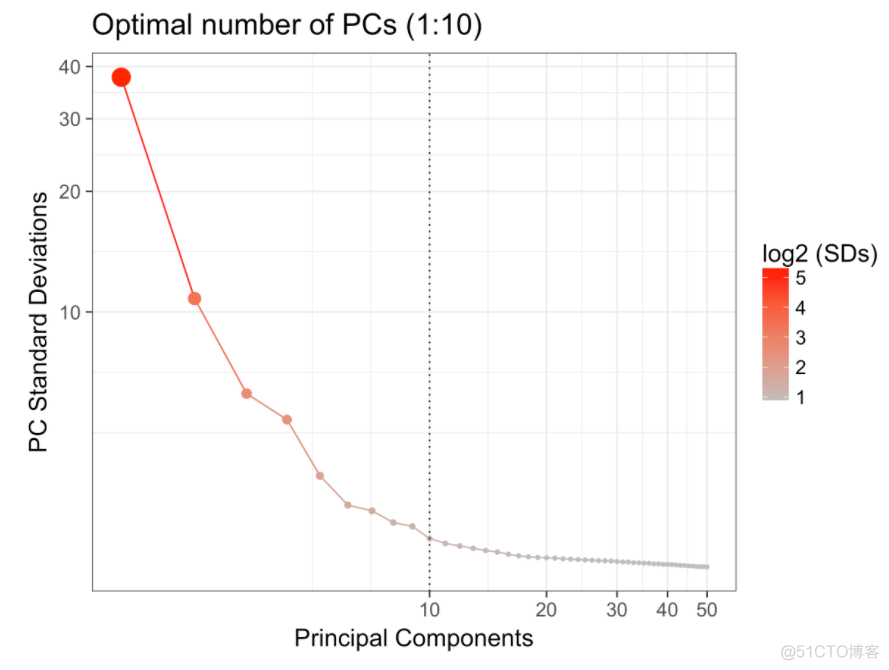
Hemberg-lab单细胞转录组数据分析(十一)- Scater单细胞表达谱PCA可视化
更多降维方法
# tSNE
my.obj 14. 对细胞进行聚类分析
“聚类是针对给定的样本,依据它们特征的相似度或距离,将其归并到若干个“类”或“簇”的数据分析问题。一个类是样本的一个子集。直观上,相似的样本聚集在相同的类,不相似的样本分散在不同的类。这里,样本之间的相似度或距离起着重要作用。”
降维是对feature (gene) 进行操作,而聚类是对sample进行操作,建议在得到高变基因以及降维后操作。(基因表达聚类分析之初探SOM)
建议使用以下默认选项:
my.obj 15. 数据可视化
# clusters
A= cluster.plot(my.obj,plot.type = "pca",interactive = F)
B= cluster.plot(my.obj,plot.type = "umap",interactive = F)
C= cluster.plot(my.obj,plot.type = "tsne",interactive = F)
D= cluster.plot(my.obj,plot.type = "diffusion",interactive = F)
#下图通过不同降维方式进行展示,其实我们可以看出,在该组数据中还是t-sne降维后的可视化表达方式最好(细胞之间的分离度较好)
library(gridExtra)
grid.arrange(A,B,C,D)
# conditions
A= cluster.plot(my.obj,plot.type = "pca",col.by = "conditions",interactive = F)
B= cluster.plot(my.obj,plot.type = "umap",col.by = "conditions",interactive = F)
C= cluster.plot(my.obj,plot.type = "tsne",col.by = "conditions",interactive = F)
D= cluster.plot(my.obj,plot.type = "diffusion",col.by = "conditions",interactive = F)
#在下图中我们可以发现无论哪种降维方式均没有batch effect不同条件下的细胞重合度较好;library(gridExtra)
grid.arrange(A,B,C,D)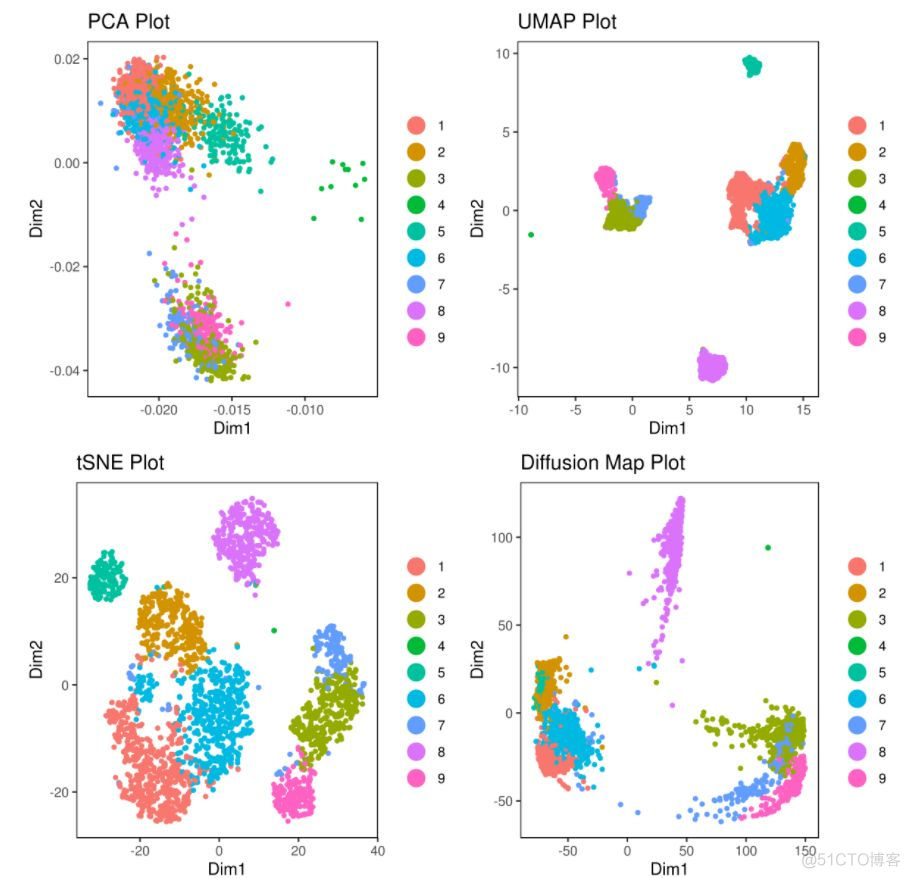

16. 三维图,密度图和交互式图
# 2D展示
cluster.plot(my.obj,
cell.size = 1,
plot.type = "tsne",
cell.color = "black",
back.col = "white",
col.by = "clusters",
cell.transparency = 0.5,
clust.dim = 2,
interactive = F)
# 可交互的2D图
cluster.plot(my.obj,
plot.type = "tsne",
col.by = "clusters",
clust.dim = 2,
interactive = T,
out.name = "tSNE_2D_clusters")
# 可交互的3D图
cluster.plot(my.obj,
plot.type = "tsne",
col.by = "clusters",
clust.dim = 3,
interactive = T,
out.name = "tSNE_3D_clusters")
# cluster的密度图
cluster.plot(my.obj,
plot.type = "pca",
col.by = "clusters",
interactive = F,
density=T)
# Density plot for conditions
cluster.plot(my.obj,
plot.type = "pca",
col.by = "conditions",
interactive = F,
density=T)
#下图为不同可视化方式,尽管方法多种多样,但个人感觉还是2D的方式展示是最为合适的。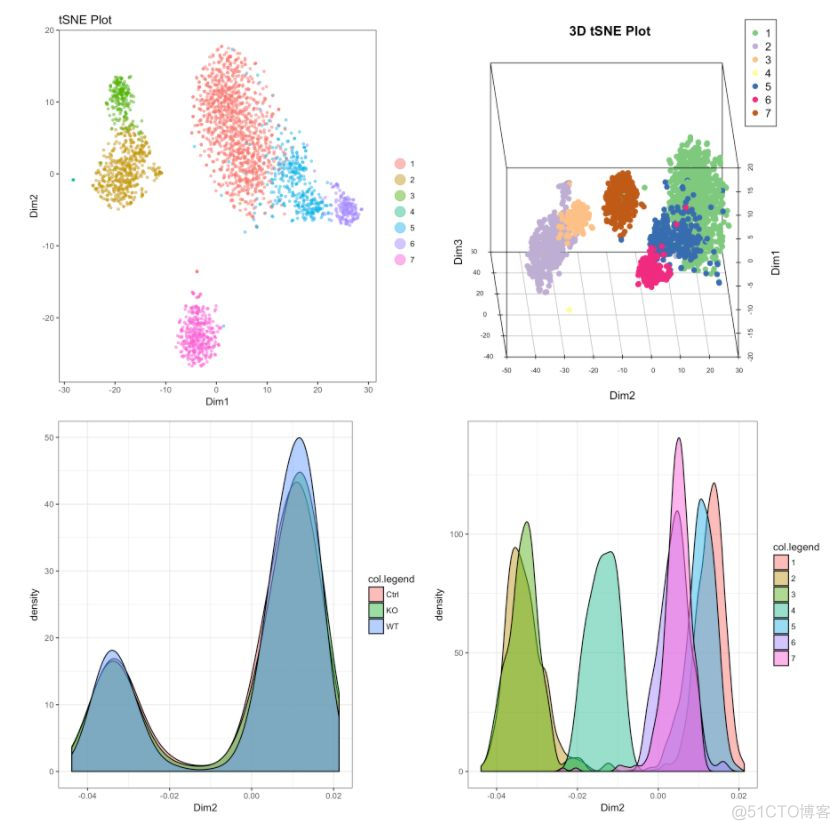
17. 不同cluster和条件下细胞比例
#如果normalize.ncell = TRUE,它将在三种状态(conditions)随机**降采样(down sample)**,因此所有条件都具有相同数量的细胞,如果为FALSE则输出原始细胞数。#下图分别是在不同条件下细胞的比例关系,KO,WT,ctrl都是我们较为常见的细胞分组。# bar plot
clust.cond.info(my.obj, plot.type = "bar", normalize.ncell = FALSE, my.out.put = "plot")
# Pie chart
clust.cond.info(my.obj, plot.type = "pie", normalize.ncell = FALSE, ,my.out.put = "plot")
# data
my.obj 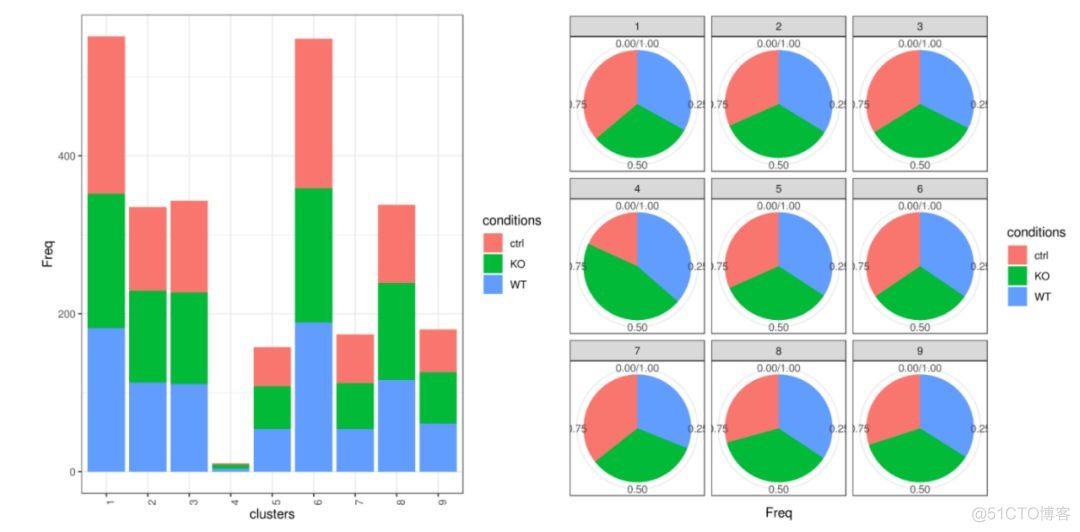
到这里,我们其实已经表现了许多的plot,下期接着介绍吧!
单细胞
- 收藏 北大生信平台” 单细胞分析、染色质分析” 视频和PPT分享
- Science: 小鼠肾脏单细胞转录组+突变分析揭示肾病潜在的细胞靶标
- Science:通过单细胞转录组测序揭示玉米减数分裂进程 | 很好的单细胞分析案例
- Nature 首次对阿尔茨海默病进行单细胞转录组分析|详细解读
- Cell 深度 一套普遍适用于各类单细胞测序数据集的锚定整合方案
- 骨髓基质在正常和白血病个体中的细胞图谱 Cell,Nature联袂解析
- 癌中之王:基质微环境塑造胰腺癌瘤内结构|Cell
- Nature系列 整合单细胞转录组学和质谱流式确定类风湿性关节炎滑膜组织中的炎症细胞状态 详细解读
- 单细胞转录组教程汇总
- 10X单细胞测序分析软件:Cell ranger,从拆库到定量
- Hemberg-lab单细胞转录组数据分析(一)- 引言
- Hemberg-lab单细胞转录组数据分析(二)- 实验平台
- Hemberg-lab单细胞转录组数据分析(三)- 原始数据质控
- Hemberg-lab单细胞转录组数据分析(四)- 文库拆分和细胞鉴定
- Hemberg-lab单细胞转录组数据分析(五)- STAR, Kallisto定量
- Hemberg-lab单细胞转录组数据分析(六)- 构建表达矩阵,UMI介绍
- Hemberg-lab单细胞转录组数据分析(七)- 导入10X和SmartSeq2数据Tabula Muris
- Hemberg-lab单细胞转录组数据分析(八)- Scater包输入导入和存储
- Hemberg-lab单细胞转录组数据分析(九)- Scater包单细胞过滤
- Hemberg-lab单细胞转录组数据分析(十)- Scater基因评估和过滤
- Hemberg-lab单细胞转录组数据分析(十一)- Scater单细胞表达谱PCA可视化
- Hemberg-lab单细胞转录组数据分析(十二)- Scater单细胞表达谱tSNE可视化
- 如何火眼金睛鉴定那些单细胞转录组中的混杂因素
- 什么?你做的差异基因方法不合适?
- 单细胞分群后,怎么找到Marker基因定义每一类群?
- 在线平台如何做单细胞测序分析全套?有它so easy!
- 植物单细胞转录组的春天来了,还不上车?Science, PC, PP, MP, bioRxiv各一个
- 三人成虎,概率却不足十分之五?
- 一文掌握GSEA,超详细教程
- 这个只需一步就可做富集分析的网站还未发表就被CNS等引用超过350次
- 什么,你算出的P-value看上去像齐天大圣变的庙?
- GO、GSEA富集分析一网打进
- GSEA富集分析 – 界面操作
- 无需写代码的高颜值富集分析神器
- 去东方,最好用的在线GO富集分析工具
- 跨物种单细胞分析发现胰腺导管癌中一类有免疫原性的抗原呈递成纤维细胞
- NCB|心咽发育多样化的单细胞转录轨迹分析
- 七龙珠|召唤一份单细胞数据库汇总
- 用了这么多年的PCA可视化竟然是错的!!!
- 单细胞预测Doublets软件包汇总-过渡态细胞是真的吗?
- Seurat亮点之细胞周期评分和回归
- cellassign:用于肿瘤微环境分析的单细胞注释工具(9月Nature)
- Nature重磅综述 |关于RNA-seq,你想知道的都在这
高颜值免费在线绘图
服务器托管,北京服务器托管,服务器租用 http://www.fwqtg.net
机房租用,北京机房租用,IDC机房托管, http://www.e1idc.net

